+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The cercosporin fungal non-reducing polyketide synthase (NR-PKS) CTB1 (SAT-KS-MAT) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Fungal non-reducing polyketide synthase (NR-PKS) / BIOSYNTHETIC PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報secondary metabolite biosynthetic process / phosphopantetheine binding / 3-oxoacyl-[acyl-carrier-protein] synthase activity / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / fatty acid biosynthetic process 類似検索 - 分子機能 | |||||||||
| 生物種 |  Cercospora nicotianae (菌類) Cercospora nicotianae (菌類) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Munoz-Hernandez H / Maier T | |||||||||
| 資金援助 | European Union,  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: The cercosporin fungal non-reducing polyketide synthase (NR-PKS) CTB1 (SAT-KS-MAT) at 2.5 Angstroms resolution 著者: Munoz-Hernandez H / Maier T | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16324.map.gz emd_16324.map.gz | 108 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16324-v30.xml emd-16324-v30.xml emd-16324.xml emd-16324.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16324.png emd_16324.png | 35.3 KB | ||
| Filedesc metadata |  emd-16324.cif.gz emd-16324.cif.gz | 6.5 KB | ||
| その他 |  emd_16324_half_map_1.map.gz emd_16324_half_map_1.map.gz emd_16324_half_map_2.map.gz emd_16324_half_map_2.map.gz | 200.5 MB 200.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16324 http://ftp.pdbj.org/pub/emdb/structures/EMD-16324 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16324 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16324 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16324_validation.pdf.gz emd_16324_validation.pdf.gz | 1019.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16324_full_validation.pdf.gz emd_16324_full_validation.pdf.gz | 1019.4 KB | 表示 | |
| XML形式データ |  emd_16324_validation.xml.gz emd_16324_validation.xml.gz | 15.7 KB | 表示 | |
| CIF形式データ |  emd_16324_validation.cif.gz emd_16324_validation.cif.gz | 18.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16324 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16324 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16324 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16324 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8by8MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16324.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16324.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
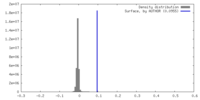
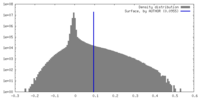
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_16324_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16324_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Non-reducing polyketide synthase CTB1 -SKM-
| 全体 | 名称: Non-reducing polyketide synthase CTB1 -SKM- |
|---|---|
| 要素 |
|
-超分子 #1: Non-reducing polyketide synthase CTB1 -SKM-
| 超分子 | 名称: Non-reducing polyketide synthase CTB1 -SKM- / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Cercospora nicotianae (菌類) Cercospora nicotianae (菌類) |
-分子 #1: Non-reducing polyketide synthase CTB1
| 分子 | 名称: Non-reducing polyketide synthase CTB1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの |
|---|---|
| 由来(天然) | 生物種:  Cercospora nicotianae (菌類) Cercospora nicotianae (菌類) |
| 分子量 | 理論値: 140.303062 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEDGAQMRVV AFGDQTYDCS EAVSQLLRVR DDAIVVDFLE RAPAVLKAEL ARLSSEQQEE TPRFATLAEL VPRYRAGTLN PAVSQALTC IAQLGLFIRQ HSSGQEAYPT AHDSCITGVC TGALTAVAVG SASSVTALVP LALHTVAVAV RLGARAWEIG S CLADARRG ...文字列: MEDGAQMRVV AFGDQTYDCS EAVSQLLRVR DDAIVVDFLE RAPAVLKAEL ARLSSEQQEE TPRFATLAEL VPRYRAGTLN PAVSQALTC IAQLGLFIRQ HSSGQEAYPT AHDSCITGVC TGALTAVAVG SASSVTALVP LALHTVAVAV RLGARAWEIG S CLADARRG ANGRYASWTS AVGGISPQDL QDRISAYTAE QALASVSVPY LSAAVGPGQS SVSAAPVILD AFLSTLLRPL TT TRLPITA PYHAPHLFTA KDVQHVTDCL PPSEAWPTVR IPIISFSRDE AVSRGASFPA AMSEAVRDCL IRPIALDRMA VSI ANHARD LGKDSVLPSP IALSFSDKLG PQVNSHLPGA KAPTPELTSK SIPSAIGAEQ QPMAKSPIAI LAASGRFPQS SSMD QFWDV LINGVDTHEL VPPTRWNAAT HVSEDPKAKN VSGTGFGCWL HEAGEFDAAY FNMSPREAPQ VDPAQRLALL TATEA LEQA GVVPNRTSST QKNRVGVWYG ATSNDWMETN SAQNVDTYFI PGGNRAFIPG RVNYFHKFSG PSYTIDTACS SSLAAL HMA CNALWRGEVD TAIVGGTNVL TNPDMTAGLD AGHFLSRSGN CKTFDDEADG YCRGEAVVTL ILKRLPDAQA DKDPIQA SI LGIATNHSAE AASITRPHAG AQQDLFQQVL TETGLTANDI SVCEMHGTGT QAGDSGETTS VVETLAPLNR SGSAVRTT P LYIGAVKSNV GHAESAAGVS SLAKILLMLK HSKIPPHVGI KTKLNHRLPD LAARNTHIAR SEVPWPRPKN GKRRVLLNN FSAAGGNTCL VLEDAPEPED SQEVDPREHH IVALSAKTPD SMVNNLTNMI TWIDKHSGDS LATLPQLSYT TTARRVHHRH RAVATGTDL LQIRSSLQEQ LDRRVSGERS IPHPPNGPSF VLAFTGQGSA FAGMGVDLYK RFASFRSDIA RYDQICEGMS L PSIKAMFE DEKVFSTASP TLQQLTHVCF QMALYRLWKS LGVQAKAVVG HSLGEYAALY AAGVLSQSDT LYLVGRRAQL ME KHLSQGT HAMLAVRAKE EAIVAAIDGP PGEAYEFSCR NGEQRNVLGG TVAQIQAAKA ALEAKKIRCQ YLDTPMAFHT GQV DPILPE LLQVAAACSI QDPQIPVISP AYGKVIRSAK DFQPEYFTHH CRSSVNMVDA LQSAVEEGLL DKNVIGLEIG PGPV VTQFV KEAVGTTMQT FASINKDKDT WQLMTQALAK FYLAGASVEW SRYHEDFPGA QKVLELPAYG WALKNYWLQY VNDWS LRKG DPAVVVAASA AALEHHHHHH UniProtKB: Non-reducing polyketide synthase CTB1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 8742 / 平均電子線量: 58.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm 最大 デフォーカス(補正後): 3.3000000000000003 µm 最小 デフォーカス(補正後): 0.6 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)