+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Double-motif Single-stranded Paranemic Crossover RNA Triangle (2PXT) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA origami / Nanostructure / self-assembly / RNA | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.56 Å | |||||||||
 データ登録者 データ登録者 | Sampedro N / Andersen ES / McRae EKS | |||||||||
| 資金援助 |  デンマーク, 1件 デンマーク, 1件
| |||||||||
 引用 引用 |  ジャーナル: Small / 年: 2023 ジャーナル: Small / 年: 2023タイトル: An RNA Paranemic Crossover Triangle as A 3D Module for Cotranscriptional Nanoassembly. 著者: Néstor Sampedro Vallina / Ewan K S McRae / Cody Geary / Ebbe Sloth Andersen /  要旨: RNA nanotechnology takes advantage of structural modularity to build self-assembling nano-architectures with applications in medicine and synthetic biology. The use of paranemic motifs, that form ...RNA nanotechnology takes advantage of structural modularity to build self-assembling nano-architectures with applications in medicine and synthetic biology. The use of paranemic motifs, that form without unfolding existing secondary structure, allows for the creation of RNA nanostructures that are compatible with cotranscriptional folding in vitro and in vivo. In previous work, kissing-loop (KL) motifs have been widely used to design RNA nanostructures that fold cotranscriptionally. However, the paranemic crossover (PX) motif has not yet been explored for cotranscriptional RNA origami architectures and information about the structural geometry of the motif is unknown. Here, a six base pair-wide paranemic RNA interaction that arranges double helices in a perpendicular manner is introduced, allowing for the generation of a new and versatile building block: the paranemic-crossover triangle (PXT). The PXT is self-assembled by cotranscriptional folding and characterized by cryogenic electron microscopy, revealing for the first time an RNA PX interaction in high structural detail. The PXT is used as a building block for the construction of multimers that form filaments and rings and a duplicated PXT motif is used as a building block to self-assemble cubic structures, demonstrating the PXT as a rigid self-folding domain for the development of wireframe RNA origami architectures. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16245.map.gz emd_16245.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16245-v30.xml emd-16245-v30.xml emd-16245.xml emd-16245.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16245.png emd_16245.png | 31.7 KB | ||
| Filedesc metadata |  emd-16245.cif.gz emd-16245.cif.gz | 5.2 KB | ||
| その他 |  emd_16245_half_map_1.map.gz emd_16245_half_map_1.map.gz emd_16245_half_map_2.map.gz emd_16245_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16245 http://ftp.pdbj.org/pub/emdb/structures/EMD-16245 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16245 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16245 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16245_validation.pdf.gz emd_16245_validation.pdf.gz | 801.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16245_full_validation.pdf.gz emd_16245_full_validation.pdf.gz | 800.8 KB | 表示 | |
| XML形式データ |  emd_16245_validation.xml.gz emd_16245_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_16245_validation.cif.gz emd_16245_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16245 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16245 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16245 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16245 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16245.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16245.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.62 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half-A
| ファイル | emd_16245_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-A | ||||||||||||
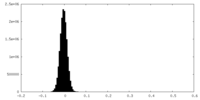
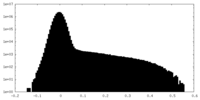
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-B
| ファイル | emd_16245_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-B | ||||||||||||
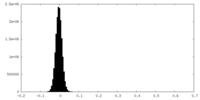
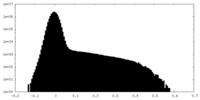
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Double-motif Single-stranded Paranemic Crossover RNA Triangle (2PXT)
| 全体 | 名称: Double-motif Single-stranded Paranemic Crossover RNA Triangle (2PXT) |
|---|---|
| 要素 |
|
-超分子 #1: Double-motif Single-stranded Paranemic Crossover RNA Triangle (2PXT)
| 超分子 | 名称: Double-motif Single-stranded Paranemic Crossover RNA Triangle (2PXT) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: In vitro transcribed RNA purified by SEC. |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 113.63 kDa/nm |
-分子 #1: RNA (354-MER)
| 分子 | 名称: RNA (354-MER) / タイプ: rna / ID: 1 / 詳細: In vitro transcribed RNA / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 113.647586 KDa |
| 配列 | 文字列: GUUACUCACC UCUUCGGAGA AAACGGUCCG AGCGUGGUUC GCCACUAAAA UACCGUUUUG GCCUGAAUGU GUCUGGUCCU UCCGCAGCC GUUUGAUGAG UUCGCUCAUC AUAGCGGGCU UACCAAACAG UUCGCUGCCA CCGUGCGGAA GCAGUUUCGA C UGAUAGAU ...文字列: GUUACUCACC UCUUCGGAGA AAACGGUCCG AGCGUGGUUC GCCACUAAAA UACCGUUUUG GCCUGAAUGU GUCUGGUCCU UCCGCAGCC GUUUGAUGAG UUCGCUCAUC AUAGCGGGCU UACCAAACAG UUCGCUGCCA CCGUGCGGAA GCAGUUUCGA C UGAUAGAU CACGGUGGGA CUAUUCGUAG UCUUUGGUGA GCUCGUUACG UUUCUUCGGA AACGAACGGC UGAUCUAUGA CC AGAUGUA UUCAGGCCGU GAGUAACGUC GCCGUACAUC UUCGGAUGUA GCGUCCCUAU UUUACAAACU UCGGUUUGGC UCG GAGGGA CGCAGCAGGG UUCGCCCUGC UCGGCGAC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 99 % / チャンバー内温度: 294 K 詳細: 3 microliter droplet, 4 second delay before blotting, 6 second blot, 0 second delay before plunging.. | ||||||||||||
| 詳細 | Sample was purified by size exclusion chromatography and concentrated in an Amicon spin concentrator. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 1.5 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.75 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 6.56 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.2.0) / 使用した粒子像数: 128507 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 3.2.0) |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X