+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Ligand-Free Structure of the decameric sulfofructose transaldolase BmSF-TAL | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | transaldolase / cryo-EM / decamer / sulfofructose / TRANSFERASE | ||||||||||||
| 機能・相同性 | 6-deoxy-6-sulfo-D-fructose transaldolase / Transaldolase/Fructose-6-phosphate aldolase, archaeal/bacterial / transaldolase activity / Transaldolase/Fructose-6-phosphate aldolase / Transaldolase/Fructose-6-phosphate aldolase / aldehyde-lyase activity / Aldolase-type TIM barrel / carbohydrate metabolic process / 6-deoxy-6-sulfo-D-fructose transaldolase 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Priestia megaterium DSM 319 (バクテリア) / Priestia megaterium DSM 319 (バクテリア) /  Bacillus aryabhattai (バクテリア) Bacillus aryabhattai (バクテリア) | ||||||||||||
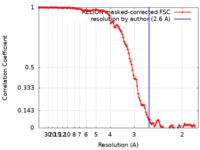
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.6 Å | ||||||||||||
 データ登録者 データ登録者 | Snow AJD / Sharma M / Blaza J / Davies GJ | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Structure and mechanism of sulfofructose transaldolase, a key enzyme in sulfoquinovose metabolism. 著者: Alexander J D Snow / Mahima Sharma / Palika Abayakoon / Spencer J Williams / James N Blaza / Gideon J Davies /   要旨: Sulfoquinovose (SQ) is a key component of plant sulfolipids (sulfoquinovosyl diacylglycerols) and a major environmental reservoir of biological sulfur. Breakdown of SQ is achieved by bacteria through ...Sulfoquinovose (SQ) is a key component of plant sulfolipids (sulfoquinovosyl diacylglycerols) and a major environmental reservoir of biological sulfur. Breakdown of SQ is achieved by bacteria through the pathways of sulfoglycolysis. The sulfoglycolytic sulfofructose transaldolase (sulfo-SFT) pathway is used by gut-resident firmicutes and soil saprophytes. After isomerization of SQ to sulfofructose (SF), the namesake enzyme catalyzes the transaldol reaction of SF transferring dihydroxyacetone to 3C/4C acceptors to give sulfolactaldehyde and fructose-6-phosphate or sedoheptulose-7-phosphate. We report the 3D cryo-EM structure of SF transaldolase from Bacillus megaterium in apo and ligand bound forms, revealing a decameric structure formed from two pentameric rings of the protomer. We demonstrate a covalent "Schiff base" intermediate formed by reaction of SF with Lys89 within a conserved Asp-Lys-Glu catalytic triad and defined by an Arg-Trp-Arg sulfonate recognition triad. The structural characterization of the signature enzyme of the sulfo-SFT pathway provides key insights into molecular recognition of the sulfonate group of sulfosugars. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15960.map.gz emd_15960.map.gz | 10.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15960-v30.xml emd-15960-v30.xml emd-15960.xml emd-15960.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15960_fsc.xml emd_15960_fsc.xml | 11.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15960.png emd_15960.png | 102.6 KB | ||
| マスクデータ |  emd_15960_msk_1.map emd_15960_msk_1.map | 144.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15960.cif.gz emd-15960.cif.gz | 6.1 KB | ||
| その他 |  emd_15960_half_map_1.map.gz emd_15960_half_map_1.map.gz emd_15960_half_map_2.map.gz emd_15960_half_map_2.map.gz | 114.5 MB 114.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15960 http://ftp.pdbj.org/pub/emdb/structures/EMD-15960 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15960 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15960 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15960_validation.pdf.gz emd_15960_validation.pdf.gz | 698.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15960_full_validation.pdf.gz emd_15960_full_validation.pdf.gz | 698.1 KB | 表示 | |
| XML形式データ |  emd_15960_validation.xml.gz emd_15960_validation.xml.gz | 19.2 KB | 表示 | |
| CIF形式データ |  emd_15960_validation.cif.gz emd_15960_validation.cif.gz | 25.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15960 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15960 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15960 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15960 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15960.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15960.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.9 Å | ||||||||||||||||||||
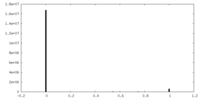
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15960_msk_1.map emd_15960_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
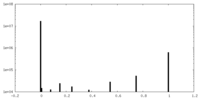
| 投影像・断面図 |
| ||||||||||||
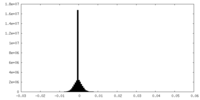
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15960_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
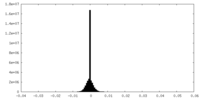
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15960_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Decameric complex of BmSF-TAL in a ligand-free state.
| 全体 | 名称: Decameric complex of BmSF-TAL in a ligand-free state. |
|---|---|
| 要素 |
|
-超分子 #1: Decameric complex of BmSF-TAL in a ligand-free state.
| 超分子 | 名称: Decameric complex of BmSF-TAL in a ligand-free state. タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Priestia megaterium DSM 319 (バクテリア) Priestia megaterium DSM 319 (バクテリア) |
-分子 #1: Transaldolase
| 分子 | 名称: Transaldolase / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO / EC番号: transaldolase |
|---|---|
| 由来(天然) | 生物種:  Bacillus aryabhattai (バクテリア) Bacillus aryabhattai (バクテリア) |
| 分子量 | 理論値: 24.443545 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKYFLDSAIL EEIRYAYENW AIDGVTTNPR HIMNSGKPFL TVLDEFASEF KGVENFPISV EINPHLDNAK DMVEEGTKIA KLSSNFVIK IPCTEPGLIA AKEFEKQGIS TNVTLVFSPS QALQPARIGA KFVSPFVGWK ENSGDDTTYI QDIVNIYKNY N YNTEIIVA ...文字列: MKYFLDSAIL EEIRYAYENW AIDGVTTNPR HIMNSGKPFL TVLDEFASEF KGVENFPISV EINPHLDNAK DMVEEGTKIA KLSSNFVIK IPCTEPGLIA AKEFEKQGIS TNVTLVFSPS QALQPARIGA KFVSPFVGWK ENSGDDTTYI QDIVNIYKNY N YNTEIIVA ALRNGKQIVD AAKAGAHIVT CGFDVYKESF QHAFTDYGLN KFRNAWDNTV UniProtKB: 6-deoxy-6-sulfo-D-fructose transaldolase |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 460 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: Buffer was clarified in a 0.22um filter and used for final SEC polishing. | |||||||||
| グリッド | モデル: Quantifoil / 材質: GOLD / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 4690 / 平均露光時間: 3.68 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 30.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X