登録情報 データベース : EMDB / ID : EMD-15757タイトル Structure of RIP2K dimer bound to the XIAP BIR2 domain cryo EM map of RIP2 kinase dimer with bound the BIR2 domain of E3 ligase XIAP 複合体 : dimeric RIP2K bound to XIAP BIR2 domain複合体 : RIP2K dimerタンパク質・ペプチド : Receptor-interacting serine/threonine-protein kinase 2複合体 : BIR2 domain of E3 ligase XIAPタンパク質・ペプチド : E3 ubiquitin-protein ligase XIAPリガンド : ZINC ION / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
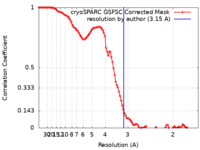
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.15 Å Pellegrini E / Cusack S 資金援助 1件 Organization Grant number 国 Not funded
ジャーナル : Life Sci Alliance / 年 : 2023タイトル : Structure shows that the BIR2 domain of E3 ligase XIAP binds across the RIPK2 kinase dimer interface.著者 : Mathilde Lethier / Karine Huard / Michael Hons / Adrien Favier / Bernhard Brutscher / Elisabetta Boeri Erba / Derek W Abbott / Stephen Cusack / Erika Pellegrini / 要旨 : RIPK2 is an essential adaptor for NOD signalling and its kinase domain is a drug target for NOD-related diseases, such as inflammatory bowel disease. However, recent work indicates that the ... RIPK2 is an essential adaptor for NOD signalling and its kinase domain is a drug target for NOD-related diseases, such as inflammatory bowel disease. However, recent work indicates that the phosphorylation activity of RIPK2 is dispensable for signalling and that inhibitors of both RIPK2 activity and RIPK2 ubiquitination prevent the essential interaction between RIPK2 and the BIR2 domain of XIAP, the key RIPK2 ubiquitin E3 ligase. Moreover, XIAP BIR2 antagonists also block this interaction. To reveal the molecular mechanisms involved, we combined native mass spectrometry, NMR, and cryo-electron microscopy to determine the structure of the RIPK2 kinase BIR2 domain complex and validated the interface with in cellulo assays. The structure shows that BIR2 binds across the RIPK2 kinase antiparallel dimer and provides an explanation for both inhibitory mechanisms. It also highlights why phosphorylation of the kinase activation loop is dispensable for signalling while revealing the structural role of RIPK2-K209 residue in the RIPK2-XIAP BIR2 interaction. Our results clarify the features of the RIPK2 conformation essential for its role as a scaffold protein for ubiquitination. 履歴 登録 2022年9月5日 - ヘッダ(付随情報) 公開 2022年10月26日 - マップ公開 2022年10月26日 - 更新 2023年9月20日 - 現状 2023年9月20日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Life Sci Alliance / 年: 2023
ジャーナル: Life Sci Alliance / 年: 2023

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_15757.map.gz
emd_15757.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-15757-v30.xml
emd-15757-v30.xml emd-15757.xml
emd-15757.xml EMDBヘッダ
EMDBヘッダ emd_15757_fsc.xml
emd_15757_fsc.xml FSCデータファイル
FSCデータファイル emd_15757.png
emd_15757.png emd_15757_msk_1.map
emd_15757_msk_1.map マスクマップ
マスクマップ emd_15757_additional_1.map.gz
emd_15757_additional_1.map.gz emd_15757_half_map_1.map.gz
emd_15757_half_map_1.map.gz emd_15757_half_map_2.map.gz
emd_15757_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-15757
http://ftp.pdbj.org/pub/emdb/structures/EMD-15757 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15757
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15757 emd_15757_validation.pdf.gz
emd_15757_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_15757_full_validation.pdf.gz
emd_15757_full_validation.pdf.gz emd_15757_validation.xml.gz
emd_15757_validation.xml.gz emd_15757_validation.cif.gz
emd_15757_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15757
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15757 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15757
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15757
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_15757.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_15757.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_15757_msk_1.map
emd_15757_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)