+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | T5 phage receptor-binding protein pb5 bound to ferrichrome transporter FhuA | |||||||||
 マップデータ マップデータ | T5 phage receptor-binding protein pb5 bound to ferrichrome transporter FhuA at 3.1 angstroms | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報siderophore transmembrane transport / siderophore uptake transmembrane transporter activity / virus tail / virion binding / toxic substance binding / transmembrane transporter complex / cell outer membrane / signaling receptor activity / intracellular iron ion homeostasis / entry receptor-mediated virion attachment to host cell ...siderophore transmembrane transport / siderophore uptake transmembrane transporter activity / virus tail / virion binding / toxic substance binding / transmembrane transporter complex / cell outer membrane / signaling receptor activity / intracellular iron ion homeostasis / entry receptor-mediated virion attachment to host cell / receptor-mediated virion attachment to host cell / symbiont entry into host cell / iron ion binding / protein domain specific binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Escherichia phage T5 (ファージ) / Escherichia phage T5 (ファージ) /  | |||||||||
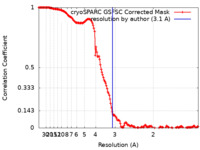
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Silale A / van den Berg B | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Structural basis for host recognition and superinfection exclusion by bacteriophage T5. 著者: Bert van den Berg / Augustinas Silale / Arnaud Baslé / Astrid F Brandner / Sophie L Mader / Syma Khalid /  要旨: A key but poorly understood stage of the bacteriophage life cycle is the binding of phage receptor-binding proteins (RBPs) to receptors on the host cell surface, leading to injection of the phage ...A key but poorly understood stage of the bacteriophage life cycle is the binding of phage receptor-binding proteins (RBPs) to receptors on the host cell surface, leading to injection of the phage genome and, for lytic phages, host cell lysis. To prevent secondary infection by the same or a closely related phage and nonproductive phage adsorption to lysed cell fragments, superinfection exclusion (SE) proteins can prevent the binding of RBPs via modulation of the host receptor structure in ways that are also unclear. Here, we present the cryogenic electron microscopy (cryo-EM) structure of the phage T5 outer membrane (OM) receptor FhuA in complex with the T5 RBP pb5, and the crystal structure of FhuA complexed to the OM SE lipoprotein Llp. Pb5 inserts four loops deeply into the extracellular lumen of FhuA and contacts the plug but does not cause any conformational changes in the receptor, supporting the view that DNA translocation does not occur through the lumen of OM channels. The FhuA-Llp structure reveals that Llp is periplasmic and binds to a nonnative conformation of the plug of FhuA, causing the inward folding of two extracellular loops via "reverse" allostery. The inward-folded loops of FhuA overlap with the pb5 binding site, explaining how Llp binding to FhuA abolishes further infection of by phage T5 and suggesting a mechanism for SE via the jamming of TonB-dependent transporters by small phage lipoproteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15229.map.gz emd_15229.map.gz | 306.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15229-v30.xml emd-15229-v30.xml emd-15229.xml emd-15229.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15229_fsc.xml emd_15229_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15229.png emd_15229.png | 45.9 KB | ||
| マスクデータ |  emd_15229_msk_1.map emd_15229_msk_1.map | 325 MB |  マスクマップ マスクマップ | |
| その他 |  emd_15229_half_map_1.map.gz emd_15229_half_map_1.map.gz emd_15229_half_map_2.map.gz emd_15229_half_map_2.map.gz | 301.5 MB 301.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15229 http://ftp.pdbj.org/pub/emdb/structures/EMD-15229 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15229 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15229 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15229_validation.pdf.gz emd_15229_validation.pdf.gz | 926.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15229_full_validation.pdf.gz emd_15229_full_validation.pdf.gz | 925.7 KB | 表示 | |
| XML形式データ |  emd_15229_validation.xml.gz emd_15229_validation.xml.gz | 23.7 KB | 表示 | |
| CIF形式データ |  emd_15229_validation.cif.gz emd_15229_validation.cif.gz | 30.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15229 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15229 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15229 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15229 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8a8cMC  8a60C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15229.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15229.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T5 phage receptor-binding protein pb5 bound to ferrichrome transporter FhuA at 3.1 angstroms | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.71 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15229_msk_1.map emd_15229_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
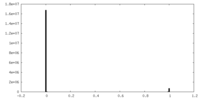
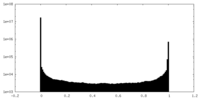
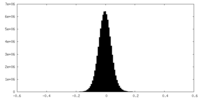
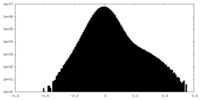
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: T5 phage receptor-binding protein pb5 bound to ferrichrome...
| ファイル | emd_15229_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T5 phage receptor-binding protein pb5 bound to ferrichrome transporter FhuA at 3.1 angstroms, half map 1 | ||||||||||||
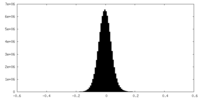
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: T5 phage receptor-binding protein pb5 bound to ferrichrome...
| ファイル | emd_15229_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | T5 phage receptor-binding protein pb5 bound to ferrichrome transporter FhuA at 3.1 angstroms, half map 2 | ||||||||||||
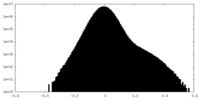
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : T5 phage receptor-binding protein pb5 bound to ferrichrome transp...
| 全体 | 名称: T5 phage receptor-binding protein pb5 bound to ferrichrome transporter FhuA |
|---|---|
| 要素 |
|
-超分子 #1: T5 phage receptor-binding protein pb5 bound to ferrichrome transp...
| 超分子 | 名称: T5 phage receptor-binding protein pb5 bound to ferrichrome transporter FhuA タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 148 KDa |
-分子 #1: Ferrichrome outer membrane transporter/phage receptor
| 分子 | 名称: Ferrichrome outer membrane transporter/phage receptor タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 78.816859 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AVEPKEDTIT VTAAPAPQES AWGPAATIAA RQSATGTKTD TPIQKVPQSI SVVTAEEMAL HQPKSVKEAL SYTPGVSVGT RGASNTYDH LIIRGFAAEG QSQNNYLNGL KLQGNFYNDA VIDPYMLERA EIMRGPVSVL YGKSSPGGLL NMVSKRPTTE P LKEVQFKA ...文字列: AVEPKEDTIT VTAAPAPQES AWGPAATIAA RQSATGTKTD TPIQKVPQSI SVVTAEEMAL HQPKSVKEAL SYTPGVSVGT RGASNTYDH LIIRGFAAEG QSQNNYLNGL KLQGNFYNDA VIDPYMLERA EIMRGPVSVL YGKSSPGGLL NMVSKRPTTE P LKEVQFKA GTDSLFQTGF DFSDSLDDDG VYSYRLTGLA RSANAQQKGS EEQRYAIAPA FTWRPDDKTN FTFLSYFQNE PE TGYYGWL PKEGTVEPLP NGKRLPTDFN EGAKNNTYSR NEKMVGYSFD HEFNDTFTVR QNLRFAENKT SQNSVYGYGV CSD PANAYS KQCAALAPAD KGHYLARKYV VDDEKLQNFS VDTQLQSKFA TGDIDHTLLT GVDFMRMRND INAWFGYDDS VPLL NLYNP VNTDFDFNAK DPANSGPYRI LNKQKQTGVY VQDQAQWDKV LVTLGGRYDW ADQESLNRVA GTTDKRDDKQ FTWRG GVNY LFDNGVTPYF SYSESFEPSS QVGKDGNIFA PSKGKQYEVG VKYVPEDRPI VVTGAVYNLT KTNNLMADPE GSFFSV EGG EIRARGVEIE AKAALSASVN VVGSYTYTDA EYTTDTTYKG NTPAQVPKHM ASLWADYTFF DGPLSGLTLG TGGRYTG SS YGDPANSFKV GSYTVVDALV RYDLARVGMA GSNVALHVNN LFDREYVASC FNTYGCFWGA ERQVVATATF RF |
-分子 #2: Receptor-binding protein pb5
| 分子 | 名称: Receptor-binding protein pb5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Escherichia phage T5 (ファージ) Escherichia phage T5 (ファージ) |
| 分子量 | 理論値: 69.611453 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSFFAGKLNN KSILSLRRGS GGDTNQHINP DSQTIFHSDM SHVIITETHS TGLRLDQGAG DYYWSEMPSR VTQLHNNDPN RVVLTEIEF SDGSRHMLSG MSMGVGAKAY GIINPQIMSQ GGLKTQITAS ADLSLDVGYF NTGTSGTIPQ KLRDGTGCQH M FGAFSGRR ...文字列: MSFFAGKLNN KSILSLRRGS GGDTNQHINP DSQTIFHSDM SHVIITETHS TGLRLDQGAG DYYWSEMPSR VTQLHNNDPN RVVLTEIEF SDGSRHMLSG MSMGVGAKAY GIINPQIMSQ GGLKTQITAS ADLSLDVGYF NTGTSGTIPQ KLRDGTGCQH M FGAFSGRR GFASSAMYLG GAALYKSAWS GSGYVVADAG TLTIPSDYVR HPGARNFGFN AIYVRGRSCN RVLYGMEGPN YT TGGAVQG ASSSGALNFT YNPSNPESPK YSVGFARADP TNYAYWESMG DPNDSANGPI GIYSEHLGIY PSKITWYVTN LVY NGSGYN IDGGLFNGND IKLSPREFII KGVNVNNTSW KFINFIEKNF NVGNRADFRD VGCNLSKDSP STGISGIATF GLPT TESNN APSIKGGNVG GLHANVVSIY NFLPSASWYV SSNPPKIGNN YGDVWSENLL PLRLLGGSGS TILSGNIVFQ GNGSV HVGT VGLDLNSSRN GAIVCTMEFI DDTWLSAGGI GCFNPTEMLS QGAEYGDSRF RIGGNTINKK LHQILSLPAG EYVPFF TIK GTVVNACKLQ AAAYNPTPYW VSGLPGSVGQ TGYYTLTYYM RNDGNNNISI WLDSSMSNII GMKACLPNIK LIIQRLT HH HHHH |
-分子 #3: [(2R,3S,4R,5R,6R)-2-[[(2R,4R,5R,6R)-6-[(1R)-1,2-bis(oxidanyl)ethy...
| 分子 | 名称: [(2R,3S,4R,5R,6R)-2-[[(2R,4R,5R,6R)-6-[(1R)-1,2-bis(oxidanyl)ethyl]-4-[(2R,4R,5R,6R)-6-[(1R)-1,2-bis(oxidanyl)ethyl]-2-carboxy-4,5-bis(oxidanyl)oxan-2-yl]oxy-2-carboxy-5-oxidanyl-oxan-2-yl] ...名称: [(2R,3S,4R,5R,6R)-2-[[(2R,4R,5R,6R)-6-[(1R)-1,2-bis(oxidanyl)ethyl]-4-[(2R,4R,5R,6R)-6-[(1R)-1,2-bis(oxidanyl)ethyl]-2-carboxy-4,5-bis(oxidanyl)oxan-2-yl]oxy-2-carboxy-5-oxidanyl-oxan-2-yl]oxymethyl]-5-[[(3R)-3-dodecanoyloxytetradecanoyl]amino]-4-(3-nonanoyloxypropanoyloxy)-6-[[(2R,3S,4R,5R,6R)-3-oxidanyl-4-[(3S)-3-oxidanyltetradecanoyl]oxy-5-[[(3R)-3-oxidanyltridecanoyl]amino]-6-phosphonatooxy-oxan-2-yl]methoxy]oxan-3-yl] phosphate タイプ: ligand / ID: 3 / コピー数: 1 / 式: LU9 |
|---|---|
| 分子量 | 理論値: 1.996235 KDa |
| Chemical component information |  ChemComp-LU9: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 8387 / 平均露光時間: 2.74 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X