+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Rabies virus glycoprotein in complex with Fab fragments of 17C7 and 1112-1 neutralizing antibodies | ||||||||||||||||||||||||
 マップデータ マップデータ | Sharpened final cryo-EM map | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | Viral Glycoprotein / Antibody / Complex / VIRAL PROTEIN | ||||||||||||||||||||||||
| 機能・相同性 | Rhabdovirus glycoprotein / Rhabdovirus spike glycoprotein / viral envelope / virion membrane / membrane / Glycoprotein 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||||||||
| 生物種 |  Rabies lyssavirus (ウイルス) / Rabies lyssavirus (ウイルス) /  Rabies virus strain Pasteur vaccin (ウイルス) / Rabies virus strain Pasteur vaccin (ウイルス) /  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||||||||||||||
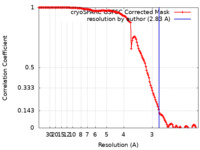
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.83 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Ng WM / Fedosyuk S / English S / Augusto G / Berg A / Thorley L / Haselon AS / Segireddy RR / Bowden TA / Douglas AD | ||||||||||||||||||||||||
| 資金援助 |  英国, European Union, 7件 英国, European Union, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2022 ジャーナル: Cell Host Microbe / 年: 2022タイトル: Structure of trimeric pre-fusion rabies virus glycoprotein in complex with two protective antibodies. 著者: Weng M Ng / Sofiya Fedosyuk / Solomon English / Gilles Augusto / Adam Berg / Luke Thorley / Anna-Sophie Haselon / Rameswara R Segireddy / Thomas A Bowden / Alexander D Douglas /  要旨: Rabies virus (RABV) causes lethal encephalitis and is responsible for approximately 60,000 deaths per year. As the sole virion-surface protein, the rabies virus glycoprotein (RABV-G) mediates host- ...Rabies virus (RABV) causes lethal encephalitis and is responsible for approximately 60,000 deaths per year. As the sole virion-surface protein, the rabies virus glycoprotein (RABV-G) mediates host-cell entry. RABV-G's pre-fusion trimeric conformation displays epitopes bound by protective neutralizing antibodies that can be induced by vaccination or passively administered for post-exposure prophylaxis. We report a 2.8-Å structure of a RABV-G trimer in the pre-fusion conformation, in complex with two neutralizing and protective monoclonal antibodies, 17C7 and 1112-1, that recognize distinct epitopes. One of these antibodies is a licensed prophylactic (17C7, Rabishield), which we show locks the protein in pre-fusion conformation. Targeted mutations can similarly stabilize RABV-G in the pre-fusion conformation, a key step toward structure-guided vaccine design. These data reveal the higher-order architecture of a key therapeutic target and the structural basis of neutralization by antibodies binding two key antigenic sites, and this will facilitate the development of improved vaccines and prophylactic antibodies. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15073.map.gz emd_15073.map.gz | 230.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15073-v30.xml emd-15073-v30.xml emd-15073.xml emd-15073.xml | 24.3 KB 24.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15073_fsc.xml emd_15073_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15073.png emd_15073.png | 123.7 KB | ||
| マスクデータ |  emd_15073_msk_1.map emd_15073_msk_1.map emd_15073_msk_2.map emd_15073_msk_2.map | 244.1 MB 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15073.cif.gz emd-15073.cif.gz | 7.2 KB | ||
| その他 |  emd_15073_half_map_1.map.gz emd_15073_half_map_1.map.gz emd_15073_half_map_2.map.gz emd_15073_half_map_2.map.gz | 226.9 MB 226.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15073 http://ftp.pdbj.org/pub/emdb/structures/EMD-15073 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15073 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15073 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15073_validation.pdf.gz emd_15073_validation.pdf.gz | 816.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15073_full_validation.pdf.gz emd_15073_full_validation.pdf.gz | 816.4 KB | 表示 | |
| XML形式データ |  emd_15073_validation.xml.gz emd_15073_validation.xml.gz | 22.4 KB | 表示 | |
| CIF形式データ |  emd_15073_validation.cif.gz emd_15073_validation.cif.gz | 29 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15073 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15073 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15073 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15073 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8a1eMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15073.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15073.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened final cryo-EM map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
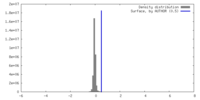
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15073_msk_1.map emd_15073_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
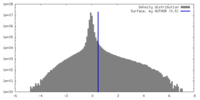
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_15073_msk_2.map emd_15073_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_15073_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
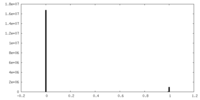
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_15073_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
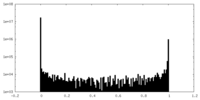
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rabies virus glycoprotein (chain A) in complex with Fab 1112-1 (c...
| 全体 | 名称: Rabies virus glycoprotein (chain A) in complex with Fab 1112-1 (chains B and C) and Fab 17C7 (chains D and E) |
|---|---|
| 要素 |
|
-超分子 #1: Rabies virus glycoprotein (chain A) in complex with Fab 1112-1 (c...
| 超分子 | 名称: Rabies virus glycoprotein (chain A) in complex with Fab 1112-1 (chains B and C) and Fab 17C7 (chains D and E) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Rabies lyssavirus (ウイルス) Rabies lyssavirus (ウイルス) |
| 分子量 | 理論値: 330 KDa |
-分子 #1: Glycoprotein
| 分子 | 名称: Glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rabies virus strain Pasteur vaccin (ウイルス) Rabies virus strain Pasteur vaccin (ウイルス)株: Pasteur vaccins / PV |
| 分子量 | 理論値: 56.494551 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: KFPIYTIPDK LGPWSPIDIH HLSCPNNLVV EDEGCTNLSG FSYMELKVGY ISAIKMNGFT CTGVVTEAET YTNFVGYVTT TFKRKHFRP TPDACRAAYN WKMAGDPRYE ESLHNPYPDY HWLRTVKTTK ESLVIISPSV ADLDPYDRSL HSPVFPGGNC S GVAVSSTY ...文字列: KFPIYTIPDK LGPWSPIDIH HLSCPNNLVV EDEGCTNLSG FSYMELKVGY ISAIKMNGFT CTGVVTEAET YTNFVGYVTT TFKRKHFRP TPDACRAAYN WKMAGDPRYE ESLHNPYPDY HWLRTVKTTK ESLVIISPSV ADLDPYDRSL HSPVFPGGNC S GVAVSSTY CSTNHDYTIW MPENPRLGMS CDIFTNSRGK RASKGSETCG FVDERGLYKS LKGACKLKLC GVLGLRLMDG TW VAMQTSN ETKWCPPGQL VNLHDFRSDE IEPLVVEELV KKREECLDAL ESIMTTKSVS FRRLSHLRKL VPGFGKAYTI FNK TLMEAD AHYKSVRTWN EIIPSKGCLR VGGRCHPHVN GVFFNGIILG PDGNVLIPEM QSSLLQQHME LLVSSVIPLM HPLA DPSTV FKNGDEAEDF VEVHLPDVHE RISGVDLGLP NWGKYVLLSA GALTALMLII FLMTCWRRVN RSEPTQHNLR GTGRE VSVT PQSGKIISSW ESHKSGGETR L UniProtKB: Glycoprotein |
-分子 #2: Fab 17C7 heavy chain variable domain
| 分子 | 名称: Fab 17C7 heavy chain variable domain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.227727 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLVESGGG VVQPGRSLRL SCAASGFTFS TYAMHWVRQA PGKGLEWVAV VSYDGRTKDY ADSVKGRFTI SRDNSKNTLY LQMNSLRTE DTAVYFCARE RFSGAYFDYW GQGTLVTVSS |
-分子 #3: Fab 17C7 light chain variable domain
| 分子 | 名称: Fab 17C7 light chain variable domain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.754044 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIVLTQSPAT LSLSPGERAT LSCRASQSVS SYLAWYQQKP GQAPRLLIYD ASNRATGIPA RFSGSGSGTD FTLTISSLEP EDFAVYSCQ QRNNWPPTFG GGTKVEIKRA |
-分子 #4: Fab 1112-1 light chain variable domain
| 分子 | 名称: Fab 1112-1 light chain variable domain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.668073 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DIVMTQSHKF MSTSIGDRVS ITCKASQDVS TAVAWYRQKP GQSPKLLIYS ASYRSTGVPD RFTGSGSGTD FTFTISSVQA EDLAVFYCQ QHYSAPLTFG AGTKLELK |
-分子 #5: Fab 1112-1 heavy chain variable domain
| 分子 | 名称: Fab 1112-1 heavy chain variable domain / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.426977 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQQPGAE LLKPGTSMKL SCKASGYTFS NYWMHWVKLR PGQGFEWIGE INPFNGGTNF NEKFKSKATL TVDRSSSTAY MQLSSVTSE DSAVYYCTIP LSDYGDWFFA VWGAGTTVTV SS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Blot for 3.5 seconds at -15 N blot force before plunging.. | |||||||||
| 詳細 | Purified sample was concentrated and mixed with 0.07% n-octyl-beta-d-glucoside to final concentrations of 1 mg/mL. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 12884 / 平均露光時間: 3.7 sec. / 平均電子線量: 44.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Model building was performed with Coot and refined with Phenix. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 108 |
| 得られたモデル |  PDB-8a1e: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)