+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
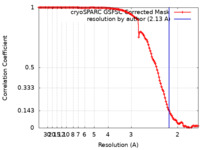
| タイトル | Cryo-EM structure of catalytically active Spinacia oleracea cytochrome b6f in complex with endogenous plastoquinones at 2.13 A resolution | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報PSII associated light-harvesting complex II binding / chloroplast photosystem I binding / chloroplast photosystem II binding / cytochrome b6f complex / plastoquinol-plastocyanin reductase / plastoquinol--plastocyanin reductase activity / electron transporter, transferring electrons within cytochrome b6/f complex of photosystem II activity / thylakoid membrane / cytochrome complex assembly / : ...PSII associated light-harvesting complex II binding / chloroplast photosystem I binding / chloroplast photosystem II binding / cytochrome b6f complex / plastoquinol-plastocyanin reductase / plastoquinol--plastocyanin reductase activity / electron transporter, transferring electrons within cytochrome b6/f complex of photosystem II activity / thylakoid membrane / cytochrome complex assembly / : / photosynthetic electron transport chain / mitochondrial electron transport, ubiquinol to cytochrome c / chloroplast thylakoid membrane / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / response to light stimulus / photosynthesis / respiratory electron transport chain / proton transmembrane transport / 2 iron, 2 sulfur cluster binding / electron transfer activity / oxidoreductase activity / iron ion binding / lipid binding / heme binding / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Spinacia oleracea (ホウレンソウ) / Spinacia oleracea (ホウレンソウ) /  spinach (ホウレンソウ) spinach (ホウレンソウ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.13 Å | |||||||||
 データ登録者 データ登録者 | Sarewicz M / Szwalec M / Pintscher S / Indyka P / Rawski M / Pietras R / Mielecki B / Koziej L / Jaciuk M / Glatt S / Osyczka A | |||||||||
| 資金援助 |  ポーランド, 2件 ポーランド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2023 ジャーナル: Sci Adv / 年: 2023タイトル: High-resolution cryo-EM structures of plant cytochrome bf at work. 著者: Marcin Sarewicz / Mateusz Szwalec / Sebastian Pintscher / Paulina Indyka / Michał Rawski / Rafał Pietras / Bohun Mielecki / Łukasz Koziej / Marcin Jaciuk / Sebastian Glatt / Artur Osyczka /  要旨: Plants use solar energy to power cellular metabolism. The oxidation of plastoquinol and reduction of plastocyanin by cytochrome bf (Cyt bf) is known as one of the key steps of photosynthesis, but the ...Plants use solar energy to power cellular metabolism. The oxidation of plastoquinol and reduction of plastocyanin by cytochrome bf (Cyt bf) is known as one of the key steps of photosynthesis, but the catalytic mechanism in the plastoquinone oxidation site (Q) remains elusive. Here, we describe two high-resolution cryo-EM structures of the spinach Cyt bf homodimer with endogenous plastoquinones and in complex with plastocyanin. Three plastoquinones are visible and line up one after another head to tail near Q in both monomers, indicating the existence of a channel in each monomer. Therefore, quinones appear to flow through Cyt bf in one direction, transiently exposing the redox-active ring of quinone during catalysis. Our work proposes an unprecedented one-way traffic model that explains efficient quinol oxidation during photosynthesis and respiration. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15027.map.gz emd_15027.map.gz | 98.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15027-v30.xml emd-15027-v30.xml emd-15027.xml emd-15027.xml | 33.2 KB 33.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15027_fsc.xml emd_15027_fsc.xml | 12.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15027.png emd_15027.png | 129.5 KB | ||
| その他 |  emd_15027_additional_1.map.gz emd_15027_additional_1.map.gz emd_15027_half_map_1.map.gz emd_15027_half_map_1.map.gz emd_15027_half_map_2.map.gz emd_15027_half_map_2.map.gz | 185.4 MB 182.4 MB 182.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15027 http://ftp.pdbj.org/pub/emdb/structures/EMD-15027 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15027 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15027 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15027_validation.pdf.gz emd_15027_validation.pdf.gz | 702.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15027_full_validation.pdf.gz emd_15027_full_validation.pdf.gz | 701.8 KB | 表示 | |
| XML形式データ |  emd_15027_validation.xml.gz emd_15027_validation.xml.gz | 20.6 KB | 表示 | |
| CIF形式データ |  emd_15027_validation.cif.gz emd_15027_validation.cif.gz | 26.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15027 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15027 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15027 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15027 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zyvMC  7qrmC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15027.map.gz / 形式: CCP4 / 大きさ: 196.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15027.map.gz / 形式: CCP4 / 大きさ: 196.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
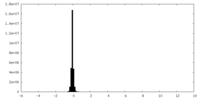
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: sharpened map
| ファイル | emd_15027_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
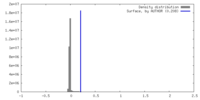
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15027_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
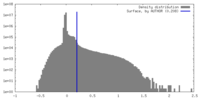
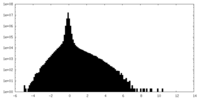
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15027_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Cytochrome b6f complex
+超分子 #1: Cytochrome b6f complex
+分子 #1: Cytochrome b6
+分子 #2: Cytochrome b6-f complex subunit 4
+分子 #3: Cytochrome f
+分子 #4: Cytochrome b6-f complex iron-sulfur subunit, chloroplastic
+分子 #5: Cytochrome b6-f complex subunit 6
+分子 #6: Cytochrome b6-f complex subunit 7
+分子 #7: Cytochrome b6-f complex subunit 5
+分子 #8: Cytochrome b6-f complex subunit 8
+分子 #9: Thylakoid soluble phosphoprotein
+分子 #10: PROTOPORPHYRIN IX CONTAINING FE
+分子 #11: HEME C
+分子 #12: CHLOROPHYLL A
+分子 #13: UNDECYL-MALTOSIDE
+分子 #14: 2,3-DIMETHYL-5-(3,7,11,15,19,23,27,31,35-NONAMETHYL-2,6,10,14,18,...
+分子 #15: 1,2-DI-O-ACYL-3-O-[6-DEOXY-6-SULFO-ALPHA-D-GLUCOPYRANOSYL]-SN-GLYCEROL
+分子 #16: FE2/S2 (INORGANIC) CLUSTER
+分子 #17: BETA-CAROTENE
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6.2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Buffer and the diluted protein sample were filtered through a 0.22 um syringe filter. Prior to freezing, protein sample was concentrated on centrifugal filter units with 50K MWCO. | ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 30.0 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot time 2 s, wait time 0 s, blot force -1. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 7651 / 平均露光時間: 1.82 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)