+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PTX3 Pentraxin Domain | |||||||||
 マップデータ マップデータ | Cryo electron microscopy derived map of the long pentraxin 3. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報(1->3)-beta-D-glucan binding / negative regulation by host of viral exo-alpha-sialidase activity / negative regulation by host of viral glycoprotein metabolic process / negative regulation of exo-alpha-sialidase activity / negative regulation of glycoprotein metabolic process / ovarian cumulus expansion / negative regulation by host of viral process /  オプソニン化 / complement component C1q complex binding / response to yeast ...(1->3)-beta-D-glucan binding / negative regulation by host of viral exo-alpha-sialidase activity / negative regulation by host of viral glycoprotein metabolic process / negative regulation of exo-alpha-sialidase activity / negative regulation of glycoprotein metabolic process / ovarian cumulus expansion / negative regulation by host of viral process / オプソニン化 / complement component C1q complex binding / response to yeast ...(1->3)-beta-D-glucan binding / negative regulation by host of viral exo-alpha-sialidase activity / negative regulation by host of viral glycoprotein metabolic process / negative regulation of exo-alpha-sialidase activity / negative regulation of glycoprotein metabolic process / ovarian cumulus expansion / negative regulation by host of viral process /  オプソニン化 / complement component C1q complex binding / response to yeast / negative regulation of viral entry into host cell / オプソニン化 / complement component C1q complex binding / response to yeast / negative regulation of viral entry into host cell /  virion binding / positive regulation of phagocytosis / extracellular matrix organization / virion binding / positive regulation of phagocytosis / extracellular matrix organization /  細胞外マトリックス / specific granule lumen / positive regulation of nitric oxide biosynthetic process / tertiary granule lumen / 細胞外マトリックス / specific granule lumen / positive regulation of nitric oxide biosynthetic process / tertiary granule lumen /  炎症 / 炎症 /  自然免疫系 / Neutrophil degranulation / 自然免疫系 / Neutrophil degranulation /  extracellular space / extracellular region / identical protein binding extracellular space / extracellular region / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
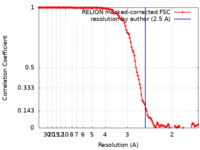
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.5 Å クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Noone DP / Sharp TH | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: PTX3 structure determination using a hybrid cryoelectron microscopy and AlphaFold approach offers insights into ligand binding and complement activation. 著者: Dylan P Noone / Douwe J Dijkstra / Teun T van der Klugt / Peter A van Veelen / Arnoud H de Ru / Paul J Hensbergen / Leendert A Trouw / Thomas H Sharp /  要旨: Pattern recognition molecules (PRMs) form an important part of innate immunity, where they facilitate the response to infections and damage by triggering processes such as inflammation. The pentraxin ...Pattern recognition molecules (PRMs) form an important part of innate immunity, where they facilitate the response to infections and damage by triggering processes such as inflammation. The pentraxin family of soluble PRMs comprises long and short pentraxins, with the former containing unique N-terminal regions unrelated to other proteins or each other. No complete high-resolution structural information exists about long pentraxins, unlike the short pentraxins, where there is an abundance of both X-ray and cryoelectron microscopy (cryo-EM)-derived structures. This study presents a high-resolution structure of the prototypical long pentraxin, PTX3. Cryo-EM yielded a 2.5-Å map of the C-terminal pentraxin domains that revealed a radically different quaternary structure compared to other pentraxins, comprising a glycosylated D4 symmetrical octameric complex stabilized by an extensive disulfide network. The cryo-EM map indicated α-helices that extended N terminal of the pentraxin domains that were not fully resolved. AlphaFold was used to predict the remaining N-terminal structure of the octameric PTX3 complex, revealing two long tetrameric coiled coils with two hinge regions, which was validated using classification of cryo-EM two-dimensional averages. The resulting hybrid cryo-EM/AlphaFold structure allowed mapping of ligand binding sites, such as C1q and fibroblast growth factor-2, as well as rationalization of previous biochemical data. Given the relevance of PTX3 in conditions ranging from COVID-19 prognosis, cancer progression, and female infertility, this structure could be used to inform the understanding and rational design of therapies for these disorders and processes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14774.map.gz emd_14774.map.gz | 12.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14774-v30.xml emd-14774-v30.xml emd-14774.xml emd-14774.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14774_fsc.xml emd_14774_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14774.png emd_14774.png | 134.2 KB | ||
| マスクデータ |  emd_14774_msk_1.map emd_14774_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| その他 |  emd_14774_additional_1.map.gz emd_14774_additional_1.map.gz emd_14774_half_map_1.map.gz emd_14774_half_map_1.map.gz emd_14774_half_map_2.map.gz emd_14774_half_map_2.map.gz | 166.1 MB 135.3 MB 135.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14774 http://ftp.pdbj.org/pub/emdb/structures/EMD-14774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14774 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zl1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14774.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14774.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo electron microscopy derived map of the long pentraxin 3. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.836 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14774_msk_1.map emd_14774_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unmasked version of the map of the long pentraxin 3.
| ファイル | emd_14774_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unmasked version of the map of the long pentraxin 3. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of the refinement job in Relion.
| ファイル | emd_14774_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of the refinement job in Relion. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of the refinement job in Relion.
| ファイル | emd_14774_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of the refinement job in Relion. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Long pentraxin 3 pentraxin domain
| 全体 | 名称: Long pentraxin 3 pentraxin domain |
|---|---|
| 要素 |
|
-超分子 #1: Long pentraxin 3 pentraxin domain
| 超分子 | 名称: Long pentraxin 3 pentraxin domain / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: CryoEM derived map of the pentraxin domains of PTX3. At higher isosurface threshold values the start of the coiled coil N-terminal domain can be seen. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 336 KDa |
-分子 #1: Pentraxin-related protein PTX3
| 分子 | 名称: Pentraxin-related protein PTX3 / タイプ: protein_or_peptide / ID: 1 詳細: Pentraxin domain of the long pentraxin 3. No N-terminal domain is included in this coordinate file. コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 41.842914 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: HHHHHHENLY FQGENSDDYD LMYVNLDNEI DNGLHPTEDP TPCACGQEHS EWDKLFIMLE NSQMRERMLL QATDDVLRGE LQRLREELG RLAESLARPC APGAPAEARL TSALDELLQA TRDAGRRLAR MEGAEAQRPE EAGRALAAVL EELRQTRADL H AVQGWAAR ...文字列: HHHHHHENLY FQGENSDDYD LMYVNLDNEI DNGLHPTEDP TPCACGQEHS EWDKLFIMLE NSQMRERMLL QATDDVLRGE LQRLREELG RLAESLARPC APGAPAEARL TSALDELLQA TRDAGRRLAR MEGAEAQRPE EAGRALAAVL EELRQTRADL H AVQGWAAR SWLPAGCETA ILFPMRSKKI FGSVHPVRPM RLESFSACIW VKATDVLNKT ILFSYGTKRN PYEIQLYLSY QS IVFVVGG EENKLVAEAM VSLGRWTHLC GTWNSEEGLT SLWVNGELAA TTVEMATGHI VPEGGILQIG QEKNGCCVGG GFD ETLAFS GRLTGFNIWD SVLSNEEIRE TGGAESCHIR GNIVGWGVTE IQPHGGAQYV S |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 8 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 379 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 23 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: HOLEY | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 65 % / チャンバー内温度: 277.15 K / 装置: LEICA EM GP | ||||||||||||
| 詳細 | PTX3 at concentrations between 1-2 uM in 20 mM Tris-HCl, 500 mM NaCl, pH 8.0 was concentrated in a 50 kDa molecular weight cut-off spin filter to concentrations between 60-70 uM. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 90.15 K / 最高: 103.15 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4612 / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)