+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | IF(heme/bound) conformation of CydDC in ADP+Pi(CydC)/ATP(CydD) bound state (Dataset-2) | |||||||||
 マップデータ マップデータ | RELION local-resolution filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ABC transporter / CydDC / Heme transporter / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cysteine export across plasma membrane / ATP-binding cassette (ABC) transporter complex, integrated substrate binding / cytochrome biosynthetic process / Translocases; Catalysing the translocation of amino acids and peptides; Linked to the hydrolysis of a nucleoside triphosphate / glutathione transmembrane transport / heme transmembrane transport / ABC-type heme transporter activity / ATPase-coupled lipid transmembrane transporter activity / ATPase-coupled transmembrane transporter activity / cell redox homeostasis ...cysteine export across plasma membrane / ATP-binding cassette (ABC) transporter complex, integrated substrate binding / cytochrome biosynthetic process / Translocases; Catalysing the translocation of amino acids and peptides; Linked to the hydrolysis of a nucleoside triphosphate / glutathione transmembrane transport / heme transmembrane transport / ABC-type heme transporter activity / ATPase-coupled lipid transmembrane transporter activity / ATPase-coupled transmembrane transporter activity / cell redox homeostasis / ATP-binding cassette (ABC) transporter complex / transmembrane transport / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
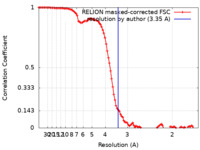
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||
 データ登録者 データ登録者 | Wu D / Safarian S | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: Dissecting the conformational complexity and mechanism of a bacterial heme transporter. 著者: Di Wu / Ahmad R Mehdipour / Franziska Finke / Hojjat G Goojani / Roan R Groh / Tamara N Grund / Thomas M B Reichhart / Rita Zimmermann / Sonja Welsch / Dirk Bald / Mark Shepherd / Gerhard ...著者: Di Wu / Ahmad R Mehdipour / Franziska Finke / Hojjat G Goojani / Roan R Groh / Tamara N Grund / Thomas M B Reichhart / Rita Zimmermann / Sonja Welsch / Dirk Bald / Mark Shepherd / Gerhard Hummer / Schara Safarian /      要旨: Iron-bound cyclic tetrapyrroles (hemes) are redox-active cofactors in bioenergetic enzymes. However, the mechanisms of heme transport and insertion into respiratory chain complexes remain unclear. ...Iron-bound cyclic tetrapyrroles (hemes) are redox-active cofactors in bioenergetic enzymes. However, the mechanisms of heme transport and insertion into respiratory chain complexes remain unclear. Here, we used cellular, biochemical, structural and computational methods to characterize the structure and function of the heterodimeric bacterial ABC transporter CydDC. We provide multi-level evidence that CydDC is a heme transporter required for functional maturation of cytochrome bd, a pharmaceutically relevant drug target. Our systematic single-particle cryogenic-electron microscopy approach combined with atomistic molecular dynamics simulations provides detailed insight into the conformational landscape of CydDC during substrate binding and occlusion. Our simulations reveal that heme binds laterally from the membrane space to the transmembrane region of CydDC, enabled by a highly asymmetrical inward-facing CydDC conformation. During the binding process, heme propionates interact with positively charged residues on the surface and later in the substrate-binding pocket of the transporter, causing the heme orientation to rotate 180°. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14640.map.gz emd_14640.map.gz | 58.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14640-v30.xml emd-14640-v30.xml emd-14640.xml emd-14640.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14640_fsc.xml emd_14640_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14640.png emd_14640.png | 75.4 KB | ||
| Filedesc metadata |  emd-14640.cif.gz emd-14640.cif.gz | 6.6 KB | ||
| その他 |  emd_14640_additional_1.map.gz emd_14640_additional_1.map.gz emd_14640_additional_2.map.gz emd_14640_additional_2.map.gz emd_14640_half_map_1.map.gz emd_14640_half_map_1.map.gz emd_14640_half_map_2.map.gz emd_14640_half_map_2.map.gz | 7.7 MB 71 MB 71.3 MB 71.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14640 http://ftp.pdbj.org/pub/emdb/structures/EMD-14640 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14640 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14640 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14640_validation.pdf.gz emd_14640_validation.pdf.gz | 881.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14640_full_validation.pdf.gz emd_14640_full_validation.pdf.gz | 881.4 KB | 表示 | |
| XML形式データ |  emd_14640_validation.xml.gz emd_14640_validation.xml.gz | 17.5 KB | 表示 | |
| CIF形式データ |  emd_14640_validation.cif.gz emd_14640_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14640 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14640 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14640 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14640 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zdbMC  7zd5C  7zdaC  7zdcC  7zdeC  7zdfC  7zdgC  7zdkC  7zdlC  7zdrC  7zdsC  7zdtC  7zduC  7zdvC  7zdwC  7ze5C  7zecC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14640.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14640.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION local-resolution filtered map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.837 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: RELION masked postprocessing map
| ファイル | emd_14640_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION masked postprocessing map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: RELION masked 3D refinement map
| ファイル | emd_14640_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION masked 3D refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: RELION 3D refinement half map 1
| ファイル | emd_14640_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION 3D refinement half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: RELION 3D refinement half map 2
| ファイル | emd_14640_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION 3D refinement half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CydDC heterodimer
| 全体 | 名称: CydDC heterodimer |
|---|---|
| 要素 |
|
-超分子 #1: CydDC heterodimer
| 超分子 | 名称: CydDC heterodimer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 130 KDa |
-分子 #1: ATP-binding/permease protein CydC
| 分子 | 名称: ATP-binding/permease protein CydC / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 62.980852 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MRALLPYLAL YKRHKWMLSL GIVLAIVTLL ASIGLLTLSG WFLSASAVAG VAGLYSFNYM LPAAGVRGAA ITRTAGRYFE RLVSHDATF RVLQHLRIYT FSKLLPLSPA GLARYRQGEL LNRVVADVDT LDHLYLRVIS PLVGAFVVIM VVTIGLSFLD F TLAFTLGG ...文字列: MRALLPYLAL YKRHKWMLSL GIVLAIVTLL ASIGLLTLSG WFLSASAVAG VAGLYSFNYM LPAAGVRGAA ITRTAGRYFE RLVSHDATF RVLQHLRIYT FSKLLPLSPA GLARYRQGEL LNRVVADVDT LDHLYLRVIS PLVGAFVVIM VVTIGLSFLD F TLAFTLGG IMLLTLFLMP PLFYRAGKST GQNLTHLRGQ YRQQLTAWLQ GQAELTIFGA SDRYRTQLEN TEIQWLEAQR RQ SELTALS QAIMLLIGAL AVILMLWMAS GGVGGNAQPG ALIALFVFCA LAAFEALAPV TGAFQHLGQV IASAVRISDL TDQ KPEVTF PDTQTRVADR VSLTLRDVQF TYPEQSQQAL KGISLQVNAG EHIAILGRTG CGKSTLLQQL TRAWDPQQGE ILLN DSPIA SLNEAALRQT ISVVPQRVHL FSATLRDNLL LASPGSSDEA LSEILRRVGL EKLLEDAGLN SWLGEGGRQL SGGEL RRLA IARALLHDAP LVLLDEPTEG LDATTESQIL ELLAEMMREK TVLMVTHRLR GLSRFQQIIV MDNGQIIEQG THAELL ARQ GRYYQFKQGL UniProtKB: Glutathione/L-cysteine transport system ATP-binding/permease protein CydC |
-分子 #2: ATP-binding/permease protein CydD
| 分子 | 名称: ATP-binding/permease protein CydD / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 65.118867 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MNKSRQKELT RWLKQQSVIS QRWLNISRLL GFVSGILIIA QAWFMARILQ HMIMENIPRE ALLLPFTLLV LTFVLRAWVV WLRERVGYH AGQHIRFAIR RQVLDRLQQA GPAWIQGKPA GSWATLVLEQ IDDMHDYYAR YLPQMALAVS VPLLIVVAIF P SNWAAALI ...文字列: MNKSRQKELT RWLKQQSVIS QRWLNISRLL GFVSGILIIA QAWFMARILQ HMIMENIPRE ALLLPFTLLV LTFVLRAWVV WLRERVGYH AGQHIRFAIR RQVLDRLQQA GPAWIQGKPA GSWATLVLEQ IDDMHDYYAR YLPQMALAVS VPLLIVVAIF P SNWAAALI LLGTAPLIPL FMALVGMGAA DANRRNFLAL ARLSGHFLDR LRGMETLRIF GRGEAEIESI RSASEDFRQR TM EVLRLAF LSSGILEFFT SLSIALVAVY FGFSYLGELD FGHYDTGVTL AAGFLALILA PEFFQPLRDL GTFYHAKAQA VGA ADSLKT FMETPLAHPQ RGEAELASTD PVTIEAEELF ITSPEGKTLA GPLNFTLPAG QRAVLVGRSG SGKSSLLNAL SGFL SYQGS LRINGIELRD LSPESWRKHL SWVGQNPQLP AATLRDNVLL ARPDASEQEL QAALDNAWVS EFLPLLPQGV DTPVG DQAA RLSVGQAQRV AVARALLNPC SLLLLDEPAA SLDAHSEQRV MEALNAASLR QTTLMVTHQL EDLADWDVIW VMQDGR IIE QGRYAELSVA GGPFATLLAH RQEEI UniProtKB: Glutathione/L-cysteine transport system ATP-binding/permease protein CydD |
-分子 #3: HEME B/C
| 分子 | 名称: HEME B/C / タイプ: ligand / ID: 3 / コピー数: 1 / 式: HEB |
|---|---|
| 分子量 | 理論値: 618.503 Da |
| Chemical component information |  ChemComp-HEB: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #6: PHOSPHITE ION
| 分子 | 名称: PHOSPHITE ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: PO3 |
|---|---|
| 分子量 | 理論値: 78.972 Da |
| Chemical component information |  ChemComp-PO3: |
-分子 #7: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 6 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 1.1 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



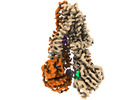






































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)