+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of POLRMT mutant. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Mitochondrial transcription initiation / mitochondrial DNA-directed RNA polymerase complex / mitochondrial promoter sequence-specific DNA binding / transcription initiation at mitochondrial promoter / mitochondrial transcription / DNA primase activity / mitochondrial nucleoid / Transcriptional activation of mitochondrial biogenesis / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase ...Mitochondrial transcription initiation / mitochondrial DNA-directed RNA polymerase complex / mitochondrial promoter sequence-specific DNA binding / transcription initiation at mitochondrial promoter / mitochondrial transcription / DNA primase activity / mitochondrial nucleoid / Transcriptional activation of mitochondrial biogenesis / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / 3'-5'-RNA exonuclease activity / sequence-specific DNA binding / mitochondrial matrix / protein-containing complex / mitochondrion / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.24 Å | |||||||||
 データ登録者 データ登録者 | Das H / Hallberg BM | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: Non-coding 7S RNA inhibits transcription via mitochondrial RNA polymerase dimerization. 著者: Xuefeng Zhu / Xie Xie / Hrishikesh Das / Benedict G Tan / Yonghong Shi / Ali Al-Behadili / Bradley Peter / Elisa Motori / Sebastian Valenzuela / Viktor Posse / Claes M Gustafsson / B Martin ...著者: Xuefeng Zhu / Xie Xie / Hrishikesh Das / Benedict G Tan / Yonghong Shi / Ali Al-Behadili / Bradley Peter / Elisa Motori / Sebastian Valenzuela / Viktor Posse / Claes M Gustafsson / B Martin Hällberg / Maria Falkenberg /   要旨: The mitochondrial genome encodes 13 components of the oxidative phosphorylation system, and altered mitochondrial transcription drives various human pathologies. A polyadenylated, non-coding RNA ...The mitochondrial genome encodes 13 components of the oxidative phosphorylation system, and altered mitochondrial transcription drives various human pathologies. A polyadenylated, non-coding RNA molecule known as 7S RNA is transcribed from a region immediately downstream of the light strand promoter in mammalian cells, and its levels change rapidly in response to physiological conditions. Here, we report that 7S RNA has a regulatory function, as it controls levels of mitochondrial transcription both in vitro and in cultured human cells. Using cryo-EM, we show that POLRMT dimerization is induced by interactions with 7S RNA. The resulting POLRMT dimer interface sequesters domains necessary for promoter recognition and unwinding, thereby preventing transcription initiation. We propose that the non-coding 7S RNA molecule is a component of a negative feedback loop that regulates mitochondrial transcription in mammalian cells. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14619.map.gz emd_14619.map.gz | 87.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14619-v30.xml emd-14619-v30.xml emd-14619.xml emd-14619.xml | 17.9 KB 17.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_14619.png emd_14619.png | 90.6 KB | ||
| その他 |  emd_14619_half_map_1.map.gz emd_14619_half_map_1.map.gz emd_14619_half_map_2.map.gz emd_14619_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14619 http://ftp.pdbj.org/pub/emdb/structures/EMD-14619 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14619 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14619 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14619_validation.pdf.gz emd_14619_validation.pdf.gz | 622.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14619_full_validation.pdf.gz emd_14619_full_validation.pdf.gz | 621.6 KB | 表示 | |
| XML形式データ |  emd_14619_validation.xml.gz emd_14619_validation.xml.gz | 12.9 KB | 表示 | |
| CIF形式データ |  emd_14619_validation.cif.gz emd_14619_validation.cif.gz | 15.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14619 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14619 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14619 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14619 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zc4MC  7pzpC  7pzrC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14619.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14619.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.01 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_14619_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
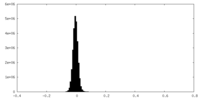
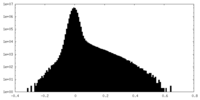
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14619_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
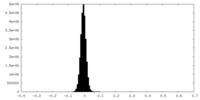
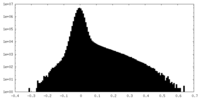
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : POLRMT
| 全体 | 名称: POLRMT |
|---|---|
| 要素 |
|
-超分子 #1: POLRMT
| 超分子 | 名称: POLRMT / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: DNA-directed RNA polymerase, mitochondrial |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 137 KDa |
-分子 #1: DNA-directed RNA polymerase, mitochondrial
| 分子 | 名称: DNA-directed RNA polymerase, mitochondrial / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 134.327703 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QSSSASPQEQ DQDRRKDWGH VELLEVLQAR VRQLQAESVS EVVVNRVDVA RLPECGSGDG SLQPPRKVQM GAKDATPVPC GRWAKILEK DKRTQQMRMQ RLKAKLQMPF QSGEFKALTR RLQVEPRLLS KQMAGCLEDC TRQAPESPWE EQLARLLQEA P GKLSLDVE ...文字列: QSSSASPQEQ DQDRRKDWGH VELLEVLQAR VRQLQAESVS EVVVNRVDVA RLPECGSGDG SLQPPRKVQM GAKDATPVPC GRWAKILEK DKRTQQMRMQ RLKAKLQMPF QSGEFKALTR RLQVEPRLLS KQMAGCLEDC TRQAPESPWE EQLARLLQEA P GKLSLDVE QAPSGQHSQA QLSGQQQRLL AFFKCCLLTD QLPLAHHLLV VHHGQAQKRA LLTLDMYNAV MLGWARQGAF KE LVYVLFM VKDAGLTPDL LSYAAALQCM GRQDQDAGTI ERCLEQMSQE GLKLQALFTA VLLSEEDRAT VLKAVHKVKP TFS LPPQLP PPVNTSKLLR DVYAKDGRVS YPKLHLPLKT LQCLFEKQLH MELASRVCVV SVEKPTLPSK EVKHARATLA TLRD QWEKA LCRALRETKN RLEREVYEGR FSLYPFLCLL DEREVVRMLL QVLQALPAQG ESFTTLAREL SARTFSRHVV QRQRV SGQV QALQNHYRKY LCLLASDAEV PEPCLPRQYW EELGAPEALR EQPWPLPVQM ELGKLLAEML VQATQMPCSL DKPHRS SRL VPVLYHVYSF RNVQQIGILK PHPAYVQLLE KAAEPTLTFE AVDVPMLCPP LPWTSPHSGA FLLSPTKLMR TVEGATQ HQ ELLETCPPTA LHGALDALTQ LGNCAWRVNG RVLDLVLQLF QAKGCPQLGV PAPPSEAPQP PEAHLPHSAA PARKAELR R ELAHCQKVAR EMHSLRAEAL YRLSLAQHLR DRVFWLPHNM DFRGRTYPCP PHFNHLGSDV ARALLEFAQG RPLGPHGLD WLKIHLVNLT GLKKREPLRK RLAFAEEVMD DILDSADQPL TGRKWWMGAE EPWQTLACCM EVANAVRASD PAAYVSHLPV HQDGSCNGL QHYAALGRDS VGAASVNLEP SDVPQDVYSG VAAQVEVFRR QDAQRGMRVA QVLEGFITRK VVKQTVMTVV Y GVTRYGGR LQIEKRLREL SDFPQEFVWE ASHYLVAQVF ASLQEMFSGT RAIQHWLTES ARLISHMGSV VEWVTPLGVP VI QPYRLDS KVKQIGGGIQ SITYTHNGDI SRAPNTAKQA NGFPPNFIHS LDSSHMMLTA LHCYRKGLTF VSVHDCYWTH AAD VSVMNQ VCREQFVRLH SEPILQDLSR FLVKRFCSEP QKILEASQLK ETLQAVPKPG AFDLEQVKRS TYFFS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 6583 / 平均露光時間: 1.5 sec. / 平均電子線量: 48.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 20.0 µm / 最小 デフォーカス(補正後): 0.3 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X