+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Dipeptide and tripeptide Permease C (DtpC) | |||||||||
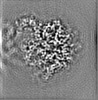
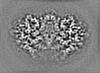
 マップデータ マップデータ | Post-processed map. Focused ref on one copy of DtpC. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Membrane protein / Peptide transporter / Proton coupled oligopeptide transporter / POT / MFS / DtpC. / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oligopeptide transport / peptide transmembrane transporter activity / membrane => GO:0016020 / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.72 Å | |||||||||
 データ登録者 データ登録者 | Killer M / Finocchio G / Pardon E / Steyaert J / Loew C | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Front Mol Biosci / 年: 2022 ジャーナル: Front Mol Biosci / 年: 2022タイトル: Cryo-EM Structure of an Atypical Proton-Coupled Peptide Transporter: Di- and Tripeptide Permease C. 著者: Maxime Killer / Giada Finocchio / Haydyn D T Mertens / Dmitri I Svergun / Els Pardon / Jan Steyaert / Christian Löw /   要旨: Proton-coupled Oligopeptide Transporters (POTs) of the Major Facilitator Superfamily (MFS) mediate the uptake of short di- and tripeptides in all phyla of life. POTs are thought to constitute the ...Proton-coupled Oligopeptide Transporters (POTs) of the Major Facilitator Superfamily (MFS) mediate the uptake of short di- and tripeptides in all phyla of life. POTs are thought to constitute the most promiscuous class of MFS transporters, with the potential to transport more than 8400 unique substrates. Over the past two decades, transport assays and biophysical studies have shown that various orthologues and paralogues display differences in substrate selectivity. The genome codes for four different POTs, known as Di- and tripeptide permeases A-D (DtpA-D). DtpC was shown previously to favor positively charged peptides as substrates. In this study, we describe, how we determined the structure of the 53 kDa DtpC by cryogenic electron microscopy (cryo-EM), and provide structural insights into the ligand specificity of this atypical POT. We collected and analyzed data on the transporter fused to split superfolder GFP (split sfGFP), in complex with a 52 kDa Pro-macrobody and with a 13 kDa nanobody. The latter sample was more stable, rigid and a significant fraction dimeric, allowing us to reconstruct a 3D volume of DtpC at a resolution of 2.7 Å. This work provides a molecular explanation for the selectivity of DtpC, and highlights the value of small and rigid fiducial markers such as nanobodies for structure determination of low molecular weight integral membrane proteins lacking soluble domains. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14618.map.gz emd_14618.map.gz | 5.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14618-v30.xml emd-14618-v30.xml emd-14618.xml emd-14618.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14618_fsc.xml emd_14618_fsc.xml | 13.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14618.png emd_14618.png | 180.7 KB | ||
| マスクデータ |  emd_14618_msk_1.map emd_14618_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14618.cif.gz emd-14618.cif.gz | 7.1 KB | ||
| その他 |  emd_14618_additional_1.map.gz emd_14618_additional_1.map.gz emd_14618_additional_2.map.gz emd_14618_additional_2.map.gz emd_14618_additional_3.map.gz emd_14618_additional_3.map.gz emd_14618_half_map_1.map.gz emd_14618_half_map_1.map.gz emd_14618_half_map_2.map.gz emd_14618_half_map_2.map.gz | 10 MB 227 MB 227 MB 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14618 http://ftp.pdbj.org/pub/emdb/structures/EMD-14618 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14618 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14618 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14618_validation.pdf.gz emd_14618_validation.pdf.gz | 900.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14618_full_validation.pdf.gz emd_14618_full_validation.pdf.gz | 899.8 KB | 表示 | |
| XML形式データ |  emd_14618_validation.xml.gz emd_14618_validation.xml.gz | 19.3 KB | 表示 | |
| CIF形式データ |  emd_14618_validation.cif.gz emd_14618_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14618 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14618 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14618 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14618 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zc2MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14618.map.gz / 形式: CCP4 / 大きさ: 5.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14618.map.gz / 形式: CCP4 / 大きさ: 5.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map. Focused ref on one copy of DtpC. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
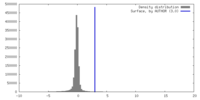
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14618_msk_1.map emd_14618_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
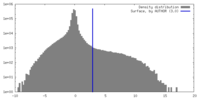
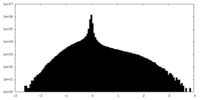
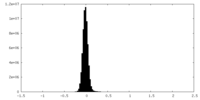
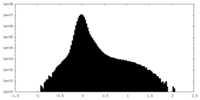
| 密度ヒストグラム |
-追加マップ: Post-processed map. Dimeric reconstruction in complex with nanobody...
| ファイル | emd_14618_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
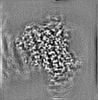
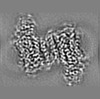
| 注釈 | Post-processed map. Dimeric reconstruction in complex with nanobody 26. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
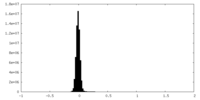
| 密度ヒストグラム |
-追加マップ: Half map 1. Dimeric reconstruction in complex with nanobody 26.
| ファイル | emd_14618_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
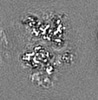
| 注釈 | Half map 1. Dimeric reconstruction in complex with nanobody 26. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
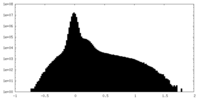
| 密度ヒストグラム |
-追加マップ: Half map 2. Dimeric reconstruction in complex with nanobody 26.
| ファイル | emd_14618_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2. Dimeric reconstruction in complex with nanobody 26. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1. Focused ref on one copy of DtpC.
| ファイル | emd_14618_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
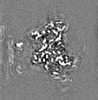
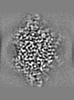
| 注釈 | Half map 1. Focused ref on one copy of DtpC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2. Focused ref on one copy of DtpC.
| ファイル | emd_14618_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
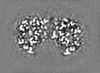
| 注釈 | Half map 2. Focused ref on one copy of DtpC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dipeptide and tripeptide permease C (DtpC)
| 全体 | 名称: Dipeptide and tripeptide permease C (DtpC) |
|---|---|
| 要素 |
|
-超分子 #1: Dipeptide and tripeptide permease C (DtpC)
| 超分子 | 名称: Dipeptide and tripeptide permease C (DtpC) / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Amino acid/peptide transporter
| 分子 | 名称: Amino acid/peptide transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.090273 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKTPSQPRAI YYIVAIQIWE YFSFYGMRAL LILYLTHQLG FDDNHAISLF SAYASLVYVT PILGGWLADR LLGNRTAVIA GALLMTLGH VVLGIDTNST FSLYLALAII ICGYGLFKSN ISCLLGELYD ENDHRRDGGF SLLYAAGNIG SIAAPIACGL A AQWYGWHV ...文字列: MKTPSQPRAI YYIVAIQIWE YFSFYGMRAL LILYLTHQLG FDDNHAISLF SAYASLVYVT PILGGWLADR LLGNRTAVIA GALLMTLGH VVLGIDTNST FSLYLALAII ICGYGLFKSN ISCLLGELYD ENDHRRDGGF SLLYAAGNIG SIAAPIACGL A AQWYGWHV GFALAGGGMF IGLLIFLSGH RHFQSTRSMD KKALTSVKFA LPVWSWLVVM LCLAPVFFTL LLENDWSGYL LA IVCLIAA QIIARMMIKF PEHRRALWQI VLLMFVGTLF WVLAQQGGST ISLFIDRFVN RQAFNIEVPT ALFQSVNAIA VML AGVVLA WLASPESRGN STLRVWLKFA FGLLLMACGF MLLAFDARHA AADGQASMGV MISGLALMGF AELFIDPVAI AQIT RLKMS GVLTGIYMLA TGAVANWLAG VVAQQTTESQ ISGMAIAAYQ RFFSQMGEWT LACVAIIVVL AFATRFLFST PTNMI QESN D UniProtKB: Amino acid/peptide transporter |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. |
| 凍結 | 凍結剤: ETHANE-PROPANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 24333 / 平均電子線量: 75.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)