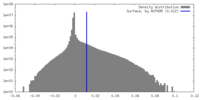
登録情報 データベース : EMDB / ID : EMD-14472タイトル Structure of the RAF1-HSP90-CDC37 complex (RHC-II) RHC-II map, local resolution filtered 複合体 : RAF1-HSP90-CDC37 complex, RHC-IIタンパク質・ペプチド : Heat shock protein HSP 90-betaタンパク質・ペプチド : RAF proto-oncogene serine/threonine-protein kinaseタンパク質・ペプチド : Hsp90 co-chaperone Cdc37リガンド : ADENOSINE-5'-TRIPHOSPHATE機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.67 Å Mesa P / Garcia-Alonso S / Barbacid M / Montoya G 資金援助 Organization Grant number 国 Novo Nordisk Foundation NNF14CC0001 Novo Nordisk Foundation NNF0024386 Novo Nordisk Foundation NNF17SA0030214 Novo Nordisk Foundation NNF18OC0055061
ジャーナル : Mol Cell / 年 : 2022タイトル : Structure of the RAF1-HSP90-CDC37 complex reveals the basis of RAF1 regulation.著者: Sara García-Alonso / Pablo Mesa / Laura de la Puente Ovejero / Gonzalo Aizpurua / Carmen G Lechuga / Eduardo Zarzuela / Clara M Santiveri / Manuel Sanclemente / Javier Muñoz / Mónica ... 著者 : Sara García-Alonso / Pablo Mesa / Laura de la Puente Ovejero / Gonzalo Aizpurua / Carmen G Lechuga / Eduardo Zarzuela / Clara M Santiveri / Manuel Sanclemente / Javier Muñoz / Mónica Musteanu / Ramón Campos-Olivas / Jorge Martínez-Torrecuadrada / Mariano Barbacid / Guillermo Montoya / 要旨 : RAF kinases are RAS-activated enzymes that initiate signaling through the MAPK cascade to control cellular proliferation, differentiation, and survival. Here, we describe the structure of the full- ... RAF kinases are RAS-activated enzymes that initiate signaling through the MAPK cascade to control cellular proliferation, differentiation, and survival. Here, we describe the structure of the full-length RAF1 protein in complex with HSP90 and CDC37 obtained by cryoelectron microscopy. The reconstruction reveals a RAF1 kinase with an unfolded N-lobe separated from its C-lobe. The hydrophobic core of the N-lobe is trapped in the HSP90 dimer, while CDC37 wraps around the chaperone and interacts with the N- and C-lobes of the kinase. The structure indicates how CDC37 can discriminate between the different members of the RAF family. Our structural analysis also reveals that the folded RAF1 assembles with 14-3-3 dimers, suggesting that after folding RAF1 follows a similar activation as B-RAF. Finally, disruption of the interaction between CDC37 and the DFG segment of RAF1 unveils potential vulnerabilities in attempting the pharmacological degradation of RAF1 for therapeutic purposes. 履歴 登録 2022年3月1日 - ヘッダ(付随情報) 公開 2022年9月14日 - マップ公開 2022年9月14日 - 更新 2022年9月28日 - 現状 2022年9月28日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 デンマーク, 4件
デンマーク, 4件  引用
引用 ジャーナル: Mol Cell / 年: 2022
ジャーナル: Mol Cell / 年: 2022

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_14472.map.gz
emd_14472.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-14472-v30.xml
emd-14472-v30.xml emd-14472.xml
emd-14472.xml EMDBヘッダ
EMDBヘッダ emd_14472_fsc.xml
emd_14472_fsc.xml FSCデータファイル
FSCデータファイル emd_14472.png
emd_14472.png emd_14472_msk_1.map
emd_14472_msk_1.map マスクマップ
マスクマップ emd_14472_half_map_1.map.gz
emd_14472_half_map_1.map.gz emd_14472_half_map_2.map.gz
emd_14472_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-14472
http://ftp.pdbj.org/pub/emdb/structures/EMD-14472 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14472
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14472 emd_14472_validation.pdf.gz
emd_14472_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_14472_full_validation.pdf.gz
emd_14472_full_validation.pdf.gz emd_14472_validation.xml.gz
emd_14472_validation.xml.gz emd_14472_validation.cif.gz
emd_14472_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14472
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14472 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14472
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14472

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_14472.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_14472.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_14472_msk_1.map
emd_14472_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト) / 組換細胞: Expi293F cells
Homo sapiens (ヒト) / 組換細胞: Expi293F cells Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー




























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)