+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Inward-facing NPA bound form of auxin transporter PIN8 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報auxin export across the plasma membrane / pollen development / auxin-activated signaling pathway / endoplasmic reticulum membrane / endoplasmic reticulum / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
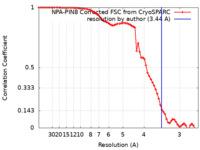
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.44 Å | |||||||||
 データ登録者 データ登録者 | Ung KL / Winkler MBL / Dedic E / Stokes DL / Pedersen BP | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Structures and mechanism of the plant PIN-FORMED auxin transporter. 著者: Kien Lam Ung / Mikael Winkler / Lukas Schulz / Martina Kolb / Dorina P Janacek / Emil Dedic / David L Stokes / Ulrich Z Hammes / Bjørn Panyella Pedersen /    要旨: Auxins are hormones that have central roles and control nearly all aspects of growth and development in plants. The proteins in the PIN-FORMED (PIN) family (also known as the auxin efflux carrier ...Auxins are hormones that have central roles and control nearly all aspects of growth and development in plants. The proteins in the PIN-FORMED (PIN) family (also known as the auxin efflux carrier family) are key participants in this process and control auxin export from the cytosol to the extracellular space. Owing to a lack of structural and biochemical data, the molecular mechanism of PIN-mediated auxin transport is not understood. Here we present biophysical analysis together with three structures of Arabidopsis thaliana PIN8: two outward-facing conformations with and without auxin, and one inward-facing conformation bound to the herbicide naphthylphthalamic acid. The structure forms a homodimer, with each monomer divided into a transport and scaffold domain with a clearly defined auxin binding site. Next to the binding site, a proline-proline crossover is a pivot point for structural changes associated with transport, which we show to be independent of proton and ion gradients and probably driven by the negative charge of the auxin. The structures and biochemical data reveal an elevator-type transport mechanism reminiscent of bile acid/sodium symporters, bicarbonate/sodium symporters and sodium/proton antiporters. Our results provide a comprehensive molecular model for auxin recognition and transport by PINs, link and expand on a well-known conceptual framework for transport, and explain a central mechanism of polar auxin transport, a core feature of plant physiology, growth and development. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14117.map.gz emd_14117.map.gz | 32.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14117-v30.xml emd-14117-v30.xml emd-14117.xml emd-14117.xml | 21.3 KB 21.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14117_fsc.xml emd_14117_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14117.png emd_14117.png | 137.6 KB | ||
| マスクデータ |  emd_14117_msk_1.map emd_14117_msk_1.map | 34.3 MB |  マスクマップ マスクマップ | |
| その他 |  emd_14117_half_map_1.map.gz emd_14117_half_map_1.map.gz emd_14117_half_map_2.map.gz emd_14117_half_map_2.map.gz | 31.7 MB 31.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14117 http://ftp.pdbj.org/pub/emdb/structures/EMD-14117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14117 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14117 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14117_validation.pdf.gz emd_14117_validation.pdf.gz | 709.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14117_full_validation.pdf.gz emd_14117_full_validation.pdf.gz | 709.1 KB | 表示 | |
| XML形式データ |  emd_14117_validation.xml.gz emd_14117_validation.xml.gz | 14.3 KB | 表示 | |
| CIF形式データ |  emd_14117_validation.cif.gz emd_14117_validation.cif.gz | 18.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14117 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14117 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14117 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14117 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7qpcMC  7qp9C  7qpaC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14117.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14117.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.294 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14117_msk_1.map emd_14117_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
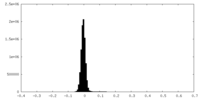
| 投影像・断面図 |
| ||||||||||||
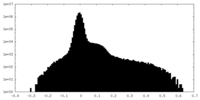
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14117_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
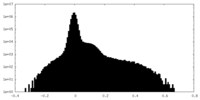
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14117_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NPA-form of PIN8
| 全体 | 名称: NPA-form of PIN8 |
|---|---|
| 要素 |
|
-超分子 #1: NPA-form of PIN8
| 超分子 | 名称: NPA-form of PIN8 / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: dimer form of PIN8 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 81 KDa |
-分子 #1: Auxin efflux carrier component 8
| 分子 | 名称: Auxin efflux carrier component 8 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.541039 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGISWLDIYH VVSATVPLYV SMTLGFLSAR HLKLFSPEQC AGINKFVAKF SIPLLSFQII SENNPFKMSP KLILSDILQK FLVVVVLAM VLRFWHPTGG RGGKLGWVIT GLSISVLPNT LILGMPILSA IYGDEAASIL EQIVVLQSLI WYTILLFLFE L NAARALPS ...文字列: MGISWLDIYH VVSATVPLYV SMTLGFLSAR HLKLFSPEQC AGINKFVAKF SIPLLSFQII SENNPFKMSP KLILSDILQK FLVVVVLAM VLRFWHPTGG RGGKLGWVIT GLSISVLPNT LILGMPILSA IYGDEAASIL EQIVVLQSLI WYTILLFLFE L NAARALPS SGASLEHTGN DQEEANIEDE PKEEEDEEEV AIVRTRSVGT MKILLKAWRK LIINPNTYAT LIGIIWATLH FR LGWNLPE MIDKSIHLLS DGGLGMAMFS LGLFMASQSS IIACGTKMAI ITMLLKFVLG PALMIASAYC IRLKSTLFKV AIL QAALPQ GVVPFVFAKE YNLHPEIIST GVIFGMLIAL PTTLAYYFLL DLPGENLYFQ |
-分子 #2: 2-(naphthalen-1-ylcarbamoyl)benzoic acid
| 分子 | 名称: 2-(naphthalen-1-ylcarbamoyl)benzoic acid / タイプ: ligand / ID: 2 / コピー数: 2 / 式: E7O |
|---|---|
| 分子量 | 理論値: 291.301 Da |
| Chemical component information |  ChemComp-E7O: |
-分子 #3: 1,2-DILINOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DILINOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: DLP |
|---|---|
| 分子量 | 理論値: 782.082 Da |
| Chemical component information | 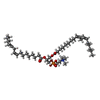 ChemComp-DLP: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 62 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 10 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 20.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 詳細: The grid was glow-discharge at 15 mA | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Wait 4 seconds after sample loading, Blotting time 4 seconds with blotting force of -1 before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / デジタル化 - サンプリング間隔: 5.0 µm / 撮影したグリッド数: 1 / 実像数: 14500 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X