| 登録情報 | データベース: EMDB / ID: EMD-14083
|
|---|
| タイトル | 26S proteasome WT-Ubp6-UbVS complex in the si state (ATPases, Rpn1, Ubp6, and UbVS) |
|---|
 マップデータ マップデータ | |
|---|
 試料 試料 | - 複合体: 26S proteasome WT-Ubp6-UbVS complex in the si state
- 複合体: yeast WT 26S proteasome body2 (ATPase, Rpn1)
- 複合体: Ubp6-UbVS
- リガンド: x 3種
|
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
proteasome regulatory particle assembly / proteasome-activating activity / proteasome regulatory particle, base subcomplex / Cross-presentation of soluble exogenous antigens (endosomes) / TNFR2 non-canonical NF-kB pathway / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Regulation of PTEN stability and activity / KEAP1-NFE2L2 pathway / CDK-mediated phosphorylation and removal of Cdc6 / Neddylation ...proteasome regulatory particle assembly / proteasome-activating activity / proteasome regulatory particle, base subcomplex / Cross-presentation of soluble exogenous antigens (endosomes) / TNFR2 non-canonical NF-kB pathway / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Regulation of PTEN stability and activity / KEAP1-NFE2L2 pathway / CDK-mediated phosphorylation and removal of Cdc6 / Neddylation / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / regulation of protein catabolic process / Orc1 removal from chromatin / MAPK6/MAPK4 signaling / proteasome storage granule / Antigen processing: Ubiquitination & Proteasome degradation / protein deubiquitination / Ub-specific processing proteases / enzyme regulator activity / Neutrophil degranulation / proteasome complex / protein-macromolecule adaptor activity / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process / ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / endoplasmic reticulum / ATP hydrolysis activity / ATP binding / nucleus / cytoplasm類似検索 - 分子機能 26S Proteasome regulatory subunit 7 / 26S Proteasome regulatory subunit 6A / 26S proteasome regulatory subunit P45-like / 26S Proteasome regulatory subunit 6B / 26S proteasome regulatory subunit 8 / Ubiquitin carboxyl-terminal hydrolase 14-like / 26S proteasome regulatory complex, non-ATPase subcomplex, Rpn1 subunit / RPN1, N-terminal / 26S proteasome non-ATPase regulatory subunit RPN1, C-terminal / RPN1 N-terminal domain ...26S Proteasome regulatory subunit 7 / 26S Proteasome regulatory subunit 6A / 26S proteasome regulatory subunit P45-like / 26S Proteasome regulatory subunit 6B / 26S proteasome regulatory subunit 8 / Ubiquitin carboxyl-terminal hydrolase 14-like / 26S proteasome regulatory complex, non-ATPase subcomplex, Rpn1 subunit / RPN1, N-terminal / 26S proteasome non-ATPase regulatory subunit RPN1, C-terminal / RPN1 N-terminal domain / 26S proteasome non-ATPase regulatory subunit RPN1 C-terminal / Proteasome/cyclosome repeat / Proteasome/cyclosome repeat / Proteasomal ATPase OB C-terminal domain / Proteasomal ATPase OB C-terminal domain / Ubiquitin specific protease (USP) domain signature 2. / Ubiquitin specific protease (USP) domain signature 1. / Ubiquitin specific protease, conserved site / Peptidase C19, ubiquitin carboxyl-terminal hydrolase / Ubiquitin carboxyl-terminal hydrolase / Ubiquitin specific protease domain / Ubiquitin specific protease (USP) domain profile. / AAA ATPase, AAA+ lid domain / AAA+ lid domain / ATPase, AAA-type, conserved site / AAA-protein family signature. / Papain-like cysteine peptidase superfamily / ATPase family associated with various cellular activities (AAA) / ATPase, AAA-type, core / : / Ubiquitin domain signature. / Ubiquitin conserved site / Ubiquitin domain / Armadillo-like helical / Ubiquitin family / Ubiquitin homologues / Ubiquitin domain profile. / Ubiquitin-like domain / Armadillo-type fold / Ubiquitin-like domain superfamily / ATPases associated with a variety of cellular activities / AAA+ ATPase domain / Nucleic acid-binding, OB-fold / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 RPT1 isoform 1 / RPT6 isoform 1 / Ubiquitin carboxyl-terminal hydrolase / RPT3 isoform 1 / RPT2 isoform 1 / RPT5 isoform 1 / RPT4 isoform 1 / Ubiquitin B / 26S proteasome regulatory subunit RPN1類似検索 - 構成要素 |
|---|
| 生物種 |   Saccharomyces cerevisiae (パン酵母) / Saccharomyces cerevisiae (パン酵母) /   baker's yeast (パン酵母) / baker's yeast (パン酵母) /  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
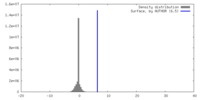
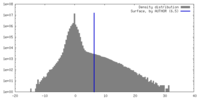
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.0 Å |
|---|
 データ登録者 データ登録者 | Hung KYS / Klumpe S / Eisele MR / Elsasser S / Geng TT / Cheng C / Joshi T / Rudack T / Sakata E / Finley D |
|---|
| 資金援助 |  ドイツ, 5件 ドイツ, 5件 | Organization | Grant number | 国 |
|---|
| German Research Foundation (DFG) | EXC 2067/1- 390729940 |  ドイツ ドイツ | | German Research Foundation (DFG) | SFB1035/Project A01 |  ドイツ ドイツ | | German Research Foundation (DFG) | CRC889/Project A11 |  ドイツ ドイツ | | Other government | Foundation for the National Institutes of Health R01 GM043601 | | | Other government | Marie Curie Career Integration grant (PCIG14-GA-2013-631577) | |
|
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022
タイトル: Allosteric control of Ubp6 and the proteasome via a bidirectional switch.
著者: Hung KYS / Klumpe S / Eisele MR / Elsasser S / Tian G / Sun S / Moroco JA / Cheng TC / Joshi T / Seibel T / Van Dalen D / Feng XH / Lu Y / Ovaa H / Engen JR / Lee BH / Rudack T / Sakata E / Finley D |
|---|
| 履歴 | | 登録 | 2021年12月23日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2022年6月1日 | - |
|---|
| マップ公開 | 2022年6月1日 | - |
|---|
| 更新 | 2022年6月1日 | - |
|---|
| 現状 | 2022年6月1日 | 処理サイト: PDBe / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報

 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 ドイツ, 5件
ドイツ, 5件  引用
引用 ジャーナル: Nat Commun / 年: 2022
ジャーナル: Nat Commun / 年: 2022 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_14083.map.gz
emd_14083.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-14083-v30.xml
emd-14083-v30.xml emd-14083.xml
emd-14083.xml EMDBヘッダ
EMDBヘッダ emd_14083.png
emd_14083.png http://ftp.pdbj.org/pub/emdb/structures/EMD-14083
http://ftp.pdbj.org/pub/emdb/structures/EMD-14083 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14083
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14083 emd_14083_validation.pdf.gz
emd_14083_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_14083_full_validation.pdf.gz
emd_14083_full_validation.pdf.gz emd_14083_validation.xml.gz
emd_14083_validation.xml.gz emd_14083_validation.cif.gz
emd_14083_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14083
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14083 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14083
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14083
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_14083.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_14083.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)