+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Tripartite ATP-independent Periplasmic (TRAP) transporter SiaQM from Photobacterium profundum in amphipol | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Photobacterium profundum (バクテリア) / Photobacterium profundum (バクテリア) /   Photobacterium profundum SS9 (バクテリア) / synthetic construct (人工物) Photobacterium profundum SS9 (バクテリア) / synthetic construct (人工物) | |||||||||
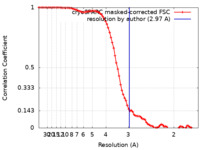
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.97 Å | |||||||||
 データ登録者 データ登録者 | North RA / Davies JS / Morado D / Dobson RCJ | |||||||||
| 資金援助 |  ニュージーランド, 2件 ニュージーランド, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure and mechanism of a tripartite ATP-independent periplasmic TRAP transporter. 著者: James S Davies / Michael J Currie / Rachel A North / Mariafrancesca Scalise / Joshua D Wright / Jack M Copping / Daniela M Remus / Ashutosh Gulati / Dustin R Morado / Sam A Jamieson / Michael ...著者: James S Davies / Michael J Currie / Rachel A North / Mariafrancesca Scalise / Joshua D Wright / Jack M Copping / Daniela M Remus / Ashutosh Gulati / Dustin R Morado / Sam A Jamieson / Michael C Newton-Vesty / Gayan S Abeysekera / Subramanian Ramaswamy / Rosmarie Friemann / Soichi Wakatsuki / Jane R Allison / Cesare Indiveri / David Drew / Peter D Mace / Renwick C J Dobson /      要旨: In bacteria and archaea, tripartite ATP-independent periplasmic (TRAP) transporters uptake essential nutrients. TRAP transporters receive their substrates via a secreted soluble substrate-binding ...In bacteria and archaea, tripartite ATP-independent periplasmic (TRAP) transporters uptake essential nutrients. TRAP transporters receive their substrates via a secreted soluble substrate-binding protein. How a sodium ion-driven secondary active transporter is strictly coupled to a substrate-binding protein is poorly understood. Here we report the cryo-EM structure of the sialic acid TRAP transporter SiaQM from Photobacterium profundum at 2.97 Å resolution. SiaM comprises a "transport" domain and a "scaffold" domain, with the transport domain consisting of helical hairpins as seen in the sodium ion-coupled elevator transporter VcINDY. The SiaQ protein forms intimate contacts with SiaM to extend the size of the scaffold domain, suggesting that TRAP transporters may operate as monomers, rather than the typically observed oligomers for elevator-type transporters. We identify the Na and sialic acid binding sites in SiaM and demonstrate a strict dependence on the substrate-binding protein SiaP for uptake. We report the SiaP crystal structure that, together with docking studies, suggest the molecular basis for how sialic acid is delivered to the SiaQM transporter complex. We thus propose a model for substrate transport by TRAP proteins, which we describe herein as an 'elevator-with-an-operator' mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13968.map.gz emd_13968.map.gz | 26.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13968-v30.xml emd-13968-v30.xml emd-13968.xml emd-13968.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13968_fsc.xml emd_13968_fsc.xml | 10 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13968.png emd_13968.png | 69.8 KB | ||
| マスクデータ |  emd_13968_msk_1.map emd_13968_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13968_half_map_1.map.gz emd_13968_half_map_1.map.gz emd_13968_half_map_2.map.gz emd_13968_half_map_2.map.gz | 48.9 MB 48.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13968 http://ftp.pdbj.org/pub/emdb/structures/EMD-13968 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13968 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13968 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13968_validation.pdf.gz emd_13968_validation.pdf.gz | 749.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13968_full_validation.pdf.gz emd_13968_full_validation.pdf.gz | 749.2 KB | 表示 | |
| XML形式データ |  emd_13968_validation.xml.gz emd_13968_validation.xml.gz | 15.3 KB | 表示 | |
| CIF形式データ |  emd_13968_validation.cif.gz emd_13968_validation.cif.gz | 19 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13968 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13968 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13968 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13968 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13968.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13968.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83063 Å | ||||||||||||||||||||||||||||||||||||
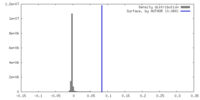
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13968_msk_1.map emd_13968_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
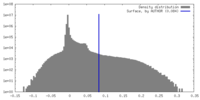
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13968_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
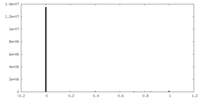
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13968_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
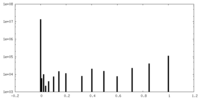
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SiaQM with megabody
| 全体 | 名称: SiaQM with megabody |
|---|---|
| 要素 |
|
-超分子 #1: SiaQM with megabody
| 超分子 | 名称: SiaQM with megabody / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-超分子 #2: SiaQM
| 超分子 | 名称: SiaQM / タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Photobacterium profundum (バクテリア) Photobacterium profundum (バクテリア) |
-超分子 #3: Megabody
| 超分子 | 名称: Megabody / タイプ: complex / ID: 3 / キメラ: Yes / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Putative TRAP-type C4-dicarboxylate transport system, small perme...
| 分子 | 名称: Putative TRAP-type C4-dicarboxylate transport system, small permease component タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Photobacterium profundum SS9 (バクテリア) / 株: SS9 Photobacterium profundum SS9 (バクテリア) / 株: SS9 |
| 分子量 | 理論値: 19.748367 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MFRKIIDNIE EIITVPLMIA LLCILTWQIS SRWLFDSPSL WSEELARVLF LHMAIIGGAI AIKKDDHVKI TFFSDKLPRN FRYSLLFAL ELLVLITIVA MIYYGYAHVQ RTAFFELITL GISSSWMTYA LPVGGCFMLV RQCQKLYFVL IDWRINENKN T SHLTACDI NE |
-分子 #2: Putative TRAP-type C4-dicarboxylate transport system, large perme...
| 分子 | 名称: Putative TRAP-type C4-dicarboxylate transport system, large permease component タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Photobacterium profundum SS9 (バクテリア) / 株: SS9 Photobacterium profundum SS9 (バクテリア) / 株: SS9 |
| 分子量 | 理論値: 45.573605 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MFTSIVGWLG LLFAGMPVGF SLIFVGLAFL VLTESTGINF AAQQMIGGLD NFTLLAVPFF VLTGHLMNSA GITERIFNFA KAMVGHITG SLGHVNILAS LLFSGMSGSA LADAGGLGQL EIKSMRDAKY DDDFAGGLTA ASCIIGPLVP PSIPLVIYGV V SNTSIGAL ...文字列: MFTSIVGWLG LLFAGMPVGF SLIFVGLAFL VLTESTGINF AAQQMIGGLD NFTLLAVPFF VLTGHLMNSA GITERIFNFA KAMVGHITG SLGHVNILAS LLFSGMSGSA LADAGGLGQL EIKSMRDAKY DDDFAGGLTA ASCIIGPLVP PSIPLVIYGV V SNTSIGAL FLAGAIPGLL CCIALCIMTY FIAKKRGYMT LPRASRKERL IAFRDAFLSL LTPFIIIGGI FSGKFTPTEA AI ISSLYAL FLGTVVYKSL TMDKFIKLVQ ETVTTTSVVA LMVMGVTVFG WIVAREQLPQ QLAELFLSIS DNPLILLLLI NLL LLFLGT FIESLALLLL LVPFLVPVAT SVGIDPVHFG VMAILNLMIG ILTPPMGMAL YVVSKVGNIP FHVLTRGVLP LLVP LFIVL GLIIVFPQIT LFLPQLVLGY GL |
-分子 #3: Megabody c7HopQ
| 分子 | 名称: Megabody c7HopQ / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 54.867098 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQESGGG LVQTKTTTSV IDTTNDAQNL LTQAQTIVNT LKDYCPILIA KSSSSNGGTN NANTPSWQTA GGGKNSCATF GAEFSAASD MINNAQKIVQ ETQQLSANQP KNITQPHNLN LNSPSSLTAL AQKMLKNAQS QAEILKLANQ VESDFNKLSS G HLKDYIGK ...文字列: QVQLQESGGG LVQTKTTTSV IDTTNDAQNL LTQAQTIVNT LKDYCPILIA KSSSSNGGTN NANTPSWQTA GGGKNSCATF GAEFSAASD MINNAQKIVQ ETQQLSANQP KNITQPHNLN LNSPSSLTAL AQKMLKNAQS QAEILKLANQ VESDFNKLSS G HLKDYIGK CDASAISSAN MTMQNQKNNW GNGCAGVEET QSLLKTSAAD FNNQTPQINQ AQNLANTLIQ ELGNNDTYEQ LS RLLTNDN GTNSKTSAQA INQAVNNLNE RAKTLAGGTT NSPAYQATLL ALRSVLGLWN SMGYAVICGG YTKSPGENNQ KDF HYTDEN GNGTTINCGG STNSNGTHSY NGTNTLKADK NVSLSIEQYE KIHEAYQILS KALKQAGLAP LNSKGEKLEA HVTT SKYAG GSLRLSCAAS GNIFDRGYMG WYRQAPGKER ELVAGISYGG STYYADSVKG RFTISRDNAK NTVYLQMNSL KPEDT AVYY CAAYPLYDDP YYYWGQGTQV TVSSLE |
-分子 #4: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 4 / コピー数: 2 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.9 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 10960 / 平均露光時間: 1.9 sec. / 平均電子線量: 71.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.4 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)