+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
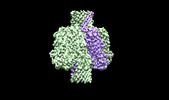
| タイトル | Cryo-EM structure of the octameric pore of Clostridium perfringens beta-toxin. | |||||||||
 マップデータ マップデータ | postprocessed map CPB | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | pore forming toxin / hemolysin / octamer / TOXIN | |||||||||
| 生物種 |   Clostridium perfringens CPE (ウェルシュ菌) Clostridium perfringens CPE (ウェルシュ菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.84 Å | |||||||||
 データ登録者 データ登録者 | Iacovache I / Zuber B | |||||||||
| 資金援助 |  スイス, 1件 スイス, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO Rep / 年: 2022 ジャーナル: EMBO Rep / 年: 2022タイトル: Cryo-EM structure of the octameric pore of Clostridium perfringens β-toxin. 著者: Julia Bruggisser / Ioan Iacovache / Samuel C Musson / Matteo T Degiacomi / Horst Posthaus / Benoît Zuber /   要旨: Clostridium perfringens is one of the most widely distributed and successful pathogens producing an impressive arsenal of toxins. One of the most potent toxins produced is the C. perfringens β-toxin ...Clostridium perfringens is one of the most widely distributed and successful pathogens producing an impressive arsenal of toxins. One of the most potent toxins produced is the C. perfringens β-toxin (CPB). This toxin is the main virulence factor of type C strains. We describe the cryo-electron microscopy (EM) structure of CPB oligomer. We show that CPB forms homo-octameric pores like the hetero-oligomeric pores of the bi-component leukocidins, with important differences in the receptor binding region and the N-terminal latch domain. Intriguingly, the octameric CPB pore complex contains a second 16-stranded β-barrel protrusion atop of the cap domain that is formed by the N-termini of the eight protomers. We propose that CPB, together with the newly identified Epx toxins, is a member a new subclass of the hemolysin-like family. In addition, we show that the β-barrel protrusion domain can be modified without affecting the pore-forming ability, thus making the pore particularly attractive for macromolecule sensing and nanotechnology. The cryo-EM structure of the octameric pore of CPB will facilitate future developments in both nanotechnology and basic research. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13876.map.gz emd_13876.map.gz | 4.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13876-v30.xml emd-13876-v30.xml emd-13876.xml emd-13876.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13876_fsc.xml emd_13876_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13876.png emd_13876.png | 47.4 KB | ||
| Filedesc metadata |  emd-13876.cif.gz emd-13876.cif.gz | 5.6 KB | ||
| その他 |  emd_13876_half_map_1.map.gz emd_13876_half_map_1.map.gz emd_13876_half_map_2.map.gz emd_13876_half_map_2.map.gz | 20.1 MB 20.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13876 http://ftp.pdbj.org/pub/emdb/structures/EMD-13876 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13876 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13876 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13876_validation.pdf.gz emd_13876_validation.pdf.gz | 649.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13876_full_validation.pdf.gz emd_13876_full_validation.pdf.gz | 649.3 KB | 表示 | |
| XML形式データ |  emd_13876_validation.xml.gz emd_13876_validation.xml.gz | 13 KB | 表示 | |
| CIF形式データ |  emd_13876_validation.cif.gz emd_13876_validation.cif.gz | 18 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13876 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13876 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13876 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13876 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13876.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13876.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocessed map CPB | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.306 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half1
| ファイル | emd_13876_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half2
| ファイル | emd_13876_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : octamer of beta toxin in SMALP
| 全体 | 名称: octamer of beta toxin in SMALP |
|---|---|
| 要素 |
|
-超分子 #1: octamer of beta toxin in SMALP
| 超分子 | 名称: octamer of beta toxin in SMALP / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 250 KDa |
-分子 #1: Clostridium perfringens beta toxin
| 分子 | 名称: Clostridium perfringens beta toxin / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Clostridium perfringens CPE (ウェルシュ菌) Clostridium perfringens CPE (ウェルシュ菌) |
| 分子量 | 理論値: 34.89375 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NDIGKTTTIT RNKTSDGYTI ITQNDKQIIS YQSVDSSSKN EDGFTASIDA RFIDDKYSSE MTTLINLTGF MSSKKEDVIK KYNLHDVTN STAINFPVRY SISILNESIN ENVKIVDSIP KNTISQKTVS NTMGYKIGGS IEIEENKPKA SIESEYAESS T IEYVQPDF ...文字列: NDIGKTTTIT RNKTSDGYTI ITQNDKQIIS YQSVDSSSKN EDGFTASIDA RFIDDKYSSE MTTLINLTGF MSSKKEDVIK KYNLHDVTN STAINFPVRY SISILNESIN ENVKIVDSIP KNTISQKTVS NTMGYKIGGS IEIEENKPKA SIESEYAESS T IEYVQPDF STIQTDHSTS KASWDTKFTE TTRGNYNLKS NNPVYGNEMF MYGRYTNVPA TENIIPDYQM SKLITGGLNP NM SVVLTAP NGTEESIIKV KMERERNCYY LNWNGANWVG QVYSRLAFDT PNVDSHIFTF KINWLTHKVT AI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 構成要素:
| ||||||
|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.25 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.8 µm |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)