+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of clamped S.cerevisiae condensin-DNA complex (Form I) | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of meiotic DNA double-strand break formation / Condensation of Prometaphase Chromosomes / meiotic chromosome condensation / tRNA gene clustering / meiotic chromosome separation / condensin complex / DNA secondary structure binding / maintenance of rDNA / rDNA chromatin condensation / synaptonemal complex assembly ...negative regulation of meiotic DNA double-strand break formation / Condensation of Prometaphase Chromosomes / meiotic chromosome condensation / tRNA gene clustering / meiotic chromosome separation / condensin complex / DNA secondary structure binding / maintenance of rDNA / rDNA chromatin condensation / synaptonemal complex assembly / nucleophagy / condensed chromosome, centromeric region / mitotic chromosome condensation / chromosome condensation / silent mating-type cassette heterochromatin formation / mitotic sister chromatid segregation / minor groove of adenine-thymine-rich DNA binding / condensed chromosome / histone binding / double-stranded DNA binding / cell division / chromatin binding / chromatin / nucleolus / ATP hydrolysis activity / mitochondrion / ATP binding / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | ||||||||||||
 データ登録者 データ登録者 | Lee B-G / Rhodes J / Lowe J | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Clamping of DNA shuts the condensin neck gate. 著者: Byung-Gil Lee / James Rhodes / Jan Löwe /  要旨: SignificanceDNA needs to be compacted to fit into nuclei and during cell division, when dense chromatids are formed for their mechanical segregation, a process that depends on the protein complex ...SignificanceDNA needs to be compacted to fit into nuclei and during cell division, when dense chromatids are formed for their mechanical segregation, a process that depends on the protein complex condensin. It forms and enlarges loops in DNA through loop extrusion. Our work resolves the atomic structure of a DNA-bound state of condensin in which ATP has not been hydrolyzed. The DNA is clamped within a compartment that has been reported previously in other structural maintenance of chromosomes (SMC) complexes, including Rad50, cohesin, and MukBEF. With the caveat of important differences, it means that all SMC complexes cycle through at least some similar states and undergo similar conformational changes in their head modules, while hydrolyzing ATP and translocating DNA. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
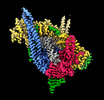
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13783.map.gz emd_13783.map.gz | 14.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13783-v30.xml emd-13783-v30.xml emd-13783.xml emd-13783.xml | 23.6 KB 23.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13783.png emd_13783.png | 126.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13783 http://ftp.pdbj.org/pub/emdb/structures/EMD-13783 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13783 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13783 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13783_validation.pdf.gz emd_13783_validation.pdf.gz | 317.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13783_full_validation.pdf.gz emd_13783_full_validation.pdf.gz | 317 KB | 表示 | |
| XML形式データ |  emd_13783_validation.xml.gz emd_13783_validation.xml.gz | 5.3 KB | 表示 | |
| CIF形式データ |  emd_13783_validation.cif.gz emd_13783_validation.cif.gz | 6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13783 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13783 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13783 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13783 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7q2xMC  7q2yC  7q2zC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13783.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13783.map.gz / 形式: CCP4 / 大きさ: 16.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||
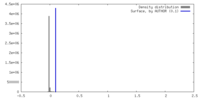
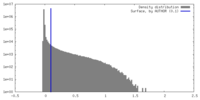
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : complex of condensin tetramer (smc2, smc4, brn1, ycs4), DNA, ADP-BeF3
+超分子 #1: complex of condensin tetramer (smc2, smc4, brn1, ycs4), DNA, ADP-BeF3
+分子 #1: Structural maintenance of chromosomes protein 2
+分子 #2: Structural maintenance of chromosomes protein 4
+分子 #3: Condensin complex subunit 2
+分子 #4: Condensin complex subunit 1
+分子 #5: DNA (36-MER)
+分子 #6: DNA (36-MER)
+分子 #7: ADENOSINE-5'-DIPHOSPHATE
+分子 #8: BERYLLIUM TRIFLUORIDE ION
+分子 #9: MAGNESIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4) |
|---|---|
| 最終 再構成 | 使用したクラス数: 1 / 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 286294 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)