+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1237 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Infectious bursal disease virus capsid assembly and maturation by structural rearrangements of a transient molecular switch. | |||||||||
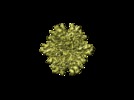
 マップデータ マップデータ | Density map of the T=1 Infectious Bursal Disease Virus (IBDV) Subviral Particle (SVP)2X2Y2Z oriented. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Infectious bursal disease virus (ウイルス) Infectious bursal disease virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 7.2 Å | |||||||||
 データ登録者 データ登録者 | Luque D / Saugar I / Rodriguez JF / Verdaguer N / Garriga D / San Martin C / Velazquez-Muriel JA / Trus BL / Carrascosa JL / Caston JR | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2007 ジャーナル: J Virol / 年: 2007タイトル: Infectious bursal disease virus capsid assembly and maturation by structural rearrangements of a transient molecular switch. 著者: Daniel Luque / Irene Saugar / José F Rodríguez / Nuria Verdaguer / Damiá Garriga / Carmen San Martín / Javier A Velázquez-Muriel / Benes L Trus / José L Carrascosa / José R Castón /  要旨: Infectious bursal disease virus (IBDV), a double-stranded RNA (dsRNA) virus belonging to the Birnaviridae family, is an economically important avian pathogen. The IBDV capsid is based on a single- ...Infectious bursal disease virus (IBDV), a double-stranded RNA (dsRNA) virus belonging to the Birnaviridae family, is an economically important avian pathogen. The IBDV capsid is based on a single-shelled T=13 lattice, and the only structural subunits are VP2 trimers. During capsid assembly, VP2 is synthesized as a protein precursor, called pVP2, whose 71-residue C-terminal end is proteolytically processed. The conformational flexibility of pVP2 is due to an amphipathic alpha-helix located at its C-terminal end. VP3, the other IBDV major structural protein that accomplishes numerous roles during the viral cycle, acts as a scaffolding protein required for assembly control. Here we address the molecular mechanism that defines the multimeric state of the capsid protein as hexamers or pentamers. We used a combination of three-dimensional cryo-electron microscopy maps at or close to subnanometer resolution with atomic models. Our studies suggest that the key polypeptide element, the C-terminal amphipathic alpha-helix, which acts as a transient conformational switch, is bound to the flexible VP2 C-terminal end. In addition, capsid protein oligomerization is also controlled by the progressive trimming of its C-terminal domain. The coordination of these molecular events correlates viral capsid assembly with different conformations of the amphipathic alpha-helix in the precursor capsid, as a five-alpha-helix bundle at the pentamers or an open star-like conformation at the hexamers. These results, reminiscent of the assembly pathway of positive single-stranded RNA viruses, such as nodavirus and tetravirus, add new insights into the evolutionary relationships of dsRNA viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1237.map.gz emd_1237.map.gz | 46.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1237-v30.xml emd-1237-v30.xml emd-1237.xml emd-1237.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1237.gif 1237.gif | 10.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1237 http://ftp.pdbj.org/pub/emdb/structures/EMD-1237 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1237 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1237 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1237.map.gz / 形式: CCP4 / 大きさ: 53.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1237.map.gz / 形式: CCP4 / 大きさ: 53.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density map of the T=1 Infectious Bursal Disease Virus (IBDV) Subviral Particle (SVP)2X2Y2Z oriented. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
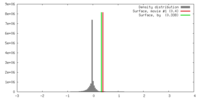
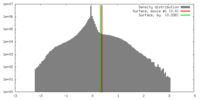
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Infectious Bursal Disease Virus Subviral Particle
| 全体 | 名称: Infectious Bursal Disease Virus Subviral Particle |
|---|---|
| 要素 |
|
-超分子 #1000: Infectious Bursal Disease Virus Subviral Particle
| 超分子 | 名称: Infectious Bursal Disease Virus Subviral Particle / タイプ: sample / ID: 1000 / 集合状態: 60 copies of VP2 arranged in 20 trimers / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 2.8 MDa |
-超分子 #1: Infectious bursal disease virus
| 超分子 | 名称: Infectious bursal disease virus / タイプ: virus / ID: 1 / Name.synonym: Gumboro virus, subviral particle / 詳細: IBDV Capsid Protein / NCBI-ID: 10995 / 生物種: Infectious bursal disease virus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes / Syn species name: Gumboro virus, subviral particle |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 実験値: 2.880 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: IBDV Subviral particle / 直径: 260 Å / T番号(三角分割数): 1 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.2 / 詳細: 25 mM PIPES, 150 mM NaCl, and 20 mM CaCl2 |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: Samples containing were applied to one side of a holey carbon film, washed twice on water drops, blotted, and plunged into a liquid ethane bath following standard procedures |
| グリッド | 詳細: 300 mesh holey carbon film |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 23 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 7.2 Å / 解像度の算出法: FSC 0.33 CUT-OFF / ソフトウェア - 名称: em3dr2 詳細: Resolution for SVP was also evaluated by FSC calculated between the full-dataset map and the SVP atomic map and for a correlation limit of 0.3, this was at 6.6 A. 使用した粒子像数: 23754 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: SITUS and URO |
| 詳細 | Protocol: Rigid body. Refinement was done in both Real and Fourier Sapce |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC and R-factor |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: SITUS and URO |
| 詳細 | Protocol: Rigid body. Refinement was done in both Real and Fourier Sapce |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: CC and R-factor |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)