+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11892 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of native royal jelly filaments | |||||||||
 マップデータ マップデータ | native royal jelly filament | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | protein filament / lipoprotein / glycosylation / royal jelly / major royal jelly protein / honeybee / PROTEIN FIBRIL | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報multicellular organism adhesion to substrate / caste determination, influence by environmental factors / rhabdomere / sterol binding / defense response to fungus / defense response to Gram-negative bacterium / killing of cells of another organism / cytoskeleton / defense response to Gram-positive bacterium / extracellular region / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Mattei S / Ban A | |||||||||
| 資金援助 | European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structure of native glycolipoprotein filaments in honeybee royal jelly. 著者: Simone Mattei / Arvid Ban / Armin Picenoni / Marc Leibundgut / Rudi Glockshuber / Daniel Boehringer /   要旨: Royal jelly (RJ) is produced by honeybees (Apis mellifera) as nutrition during larval development. The high viscosity of RJ originates from high concentrations of long lipoprotein filaments that ...Royal jelly (RJ) is produced by honeybees (Apis mellifera) as nutrition during larval development. The high viscosity of RJ originates from high concentrations of long lipoprotein filaments that include the glycosylated major royal jelly protein 1 (MRJP1), the small protein apisimin and insect lipids. Using cryo-electron microscopy we reveal the architecture and the composition of RJ filaments, in which the MRJP1 forms the outer shell of the assembly, surrounding stacked apisimin tetramers harbouring tightly packed lipids in the centre. The structural data rationalize the pH-dependent disassembly of RJ filaments in the gut of the larvae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11892.map.gz emd_11892.map.gz | 163.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11892-v30.xml emd-11892-v30.xml emd-11892.xml emd-11892.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11892.png emd_11892.png | 264.8 KB | ||
| マスクデータ |  emd_11892_msk_1.map emd_11892_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11892.cif.gz emd-11892.cif.gz | 5.9 KB | ||
| その他 |  emd_11892_half_map_1.map.gz emd_11892_half_map_1.map.gz emd_11892_half_map_2.map.gz emd_11892_half_map_2.map.gz | 138.8 MB 138.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11892 http://ftp.pdbj.org/pub/emdb/structures/EMD-11892 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11892 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11892 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11892.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11892.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | native royal jelly filament | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.19467 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
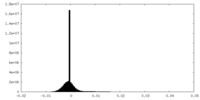
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11892_msk_1.map emd_11892_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
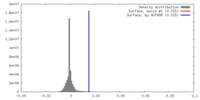
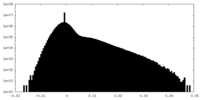
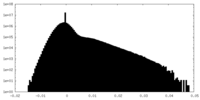
| 密度ヒストグラム |
-ハーフマップ: native royal jelly filament
| ファイル | emd_11892_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | native royal jelly filament | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
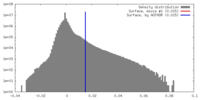
| 密度ヒストグラム |
-ハーフマップ: native royal jelly filament
| ファイル | emd_11892_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | native royal jelly filament | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : native royal jelly filaments
| 全体 | 名称: native royal jelly filaments |
|---|---|
| 要素 |
|
-超分子 #1: native royal jelly filaments
| 超分子 | 名称: native royal jelly filaments / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Major royal jelly protein 1
| 分子 | 名称: Major royal jelly protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 48.934898 KDa |
| 配列 | 文字列: MTRLFMLVCL GIVCQGTTGN ILRGESLNKS LPILHEWKFF DYDFGSDERR QDAILSGEYD YKNNYPSDID QWHDKIFVTM LRYNGVPSS LNVISKKVGD GGPLLQPYPD WSFAKYDDCS GIVSASKLAI DKCDRLWVLD SGLVNNTQPM CSPKLLTFDL T TSQLLKQV ...文字列: MTRLFMLVCL GIVCQGTTGN ILRGESLNKS LPILHEWKFF DYDFGSDERR QDAILSGEYD YKNNYPSDID QWHDKIFVTM LRYNGVPSS LNVISKKVGD GGPLLQPYPD WSFAKYDDCS GIVSASKLAI DKCDRLWVLD SGLVNNTQPM CSPKLLTFDL T TSQLLKQV EIPHDVAVNA TTGKGRLSSL AVQSLDCNTN SDTMVYIADE KGEGLIVYHN SDDSFHRLTS NTFDYDPKFT KM TIDGESY TAQDGISGMA LSPMTNNLYY SPVASTSLYY VNTEQFRTSD YQQNDIHYEG VQNILDTQSS AKVVSKSGVL FFG LVGDSA LGCWNEHRTL ERHNIRTVAQ SDETLQMIAS MKIKEALPHV PIFDRYINRE YILVLSNKMQ KMVNNDFNFD DVNF RIMNA NVNELILNTR CENPDNDRTP FKISIHL UniProtKB: Major royal jelly protein 1 |
-分子 #2: Apisimin
| 分子 | 名称: Apisimin / タイプ: protein_or_peptide / ID: 2 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.949325 KDa |
| 配列 | 文字列: MSKIVAVVVL AAFCVAMLVS DVSAKTSISV KGESNVDVVS QINSLVSSIV SGANVSAVLL AQTLVNILQI LIDANVFA UniProtKB: Apisimin |
-分子 #5: (3beta,14beta,17alpha)-ergosta-5,24(28)-dien-3-ol
| 分子 | 名称: (3beta,14beta,17alpha)-ergosta-5,24(28)-dien-3-ol / タイプ: ligand / ID: 5 / コピー数: 16 / 式: 94R |
|---|---|
| 分子量 | 理論値: 398.664 Da |
| Chemical component information | 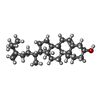 ChemComp-94R: |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 8 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #7: SULFATE ION
| 分子 | 名称: SULFATE ION / タイプ: ligand / ID: 7 / コピー数: 8 / 式: SO4 |
|---|---|
| 分子量 | 理論値: 96.063 Da |
| Chemical component information |  ChemComp-SO4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 1.7 sec. / 平均電子線量: 82.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 119050 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 105000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 54.0 Å 想定した対称性 - らせんパラメータ - ΔΦ: 64 ° 想定した対称性 - らせんパラメータ - 軸対称性: D2 (2回x2回 2面回転対称) アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0) / 使用した粒子像数: 240483 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER / 詳細: featureless cylindrical density |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: RELION |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)