+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11618 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Class 1 of three-dimensional classification of single asymmetric units of RV-A89 into six classes. The class averages reveal differences at the RNA/protein interfaces. | |||||||||
 マップデータ マップデータ | Class1 from 3D-classification of single asymmetric units of RV-A89 | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Rhinovirus A89 (ライノウイルス) Rhinovirus A89 (ライノウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Blaas D | |||||||||
| 資金援助 |  オーストリア, 2件 オーストリア, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2020 ジャーナル: Commun Biol / 年: 2020タイトル: Individual subunits of a rhinovirus causing common cold exhibit largely different protein-RNA contact site conformations. 著者: Dieter Blaas /  要旨: Rhinoviruses cause the common cold. They are icosahedral, built from sixty copies each of the capsid proteins VP1 through VP4 arranged in a pseudo T = 3 lattice. This shell encases a ss(+) RNA ...Rhinoviruses cause the common cold. They are icosahedral, built from sixty copies each of the capsid proteins VP1 through VP4 arranged in a pseudo T = 3 lattice. This shell encases a ss(+) RNA genome. Three-D classification of single and oligomeric asymmetric units computationally excised from a 2.9 Å cryo-EM density map of rhinovirus A89, showed that VP4 and the N-terminal extension of VP1 adopt different conformations within the otherwise identical 3D-structures. Analysis of up to sixty classes of single subunits and of six classes of subunit dimers, trimers, and pentamers revealed different orientations of the amino acid residues at the interface with the RNA suggesting that local asymmetry is dictated by disparities of the interacting nucleotide sequences. The different conformations escape detection by 3-D structure determination of entire virions with the conformational heterogeneity being only indicated by low density. My results do not exclude that the RNA follows a conserved assembly mechanism, contacting most or all asymmetric units in a specific way. However, as suggested by the gradual loss of asymmetry with increasing oligomerization and the 3D-structure of entire virions reconstructed by using Euler angles selected in the classification of single subunits, RNA path and/or folding likely differ from virion to virion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11618.map.gz emd_11618.map.gz | 3.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11618-v30.xml emd-11618-v30.xml emd-11618.xml emd-11618.xml | 10.7 KB 10.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11618.png emd_11618.png | 206.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11618 http://ftp.pdbj.org/pub/emdb/structures/EMD-11618 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11618 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11618 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11618_validation.pdf.gz emd_11618_validation.pdf.gz | 196.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11618_full_validation.pdf.gz emd_11618_full_validation.pdf.gz | 195.8 KB | 表示 | |
| XML形式データ |  emd_11618_validation.xml.gz emd_11618_validation.xml.gz | 7.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11618 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11618 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11618 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11618 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11618.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11618.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Class1 from 3D-classification of single asymmetric units of RV-A89 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.97 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
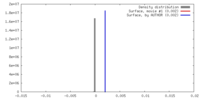
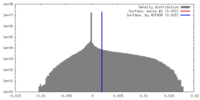
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rhinovirus A89
| 全体 | 名称:  Rhinovirus A89 (ライノウイルス) Rhinovirus A89 (ライノウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Rhinovirus A89
| 超分子 | 名称: Rhinovirus A89 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / NCBI-ID: 650130 / 生物種: Rhinovirus A89 / Sci species strain: Rhinovirus A89 / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| ウイルス殻 | Shell ID: 1 / 名称: VP1-4 / 直径: 302.0 Å |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素:
| ||||||
|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 0.4 nm | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-25 / 実像数: 5370 / 平均露光時間: 0.2 sec. / 平均電子線量: 1.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4.1) |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 107054 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 詳細 | Rosetta: DiMaio, F. et al. Atomic-accuracy models from 4.5-A cryo-electron microscopy data with density-guided iterative local refinement. Nat. Meth (2015). doi:10.1038/nmeth.3286 |
| 精密化 | 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)