+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11585 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
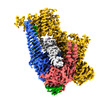
| タイトル | Cryo-EM structure of S.cerevisiae cohesin-Scc2-DNA complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SMC / cohesin / DNA / CELL CYCLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SMC loading complex / Scc2-Scc4 cohesin loading complex / mitotic cohesin loading / 2-micrometer circle DNA / Establishment of Sister Chromatid Cohesion / Resolution of Sister Chromatid Cohesion / tRNA gene clustering / meiotic cohesin complex / cohesin loader activity / establishment of meiotic sister chromatid cohesion ...SMC loading complex / Scc2-Scc4 cohesin loading complex / mitotic cohesin loading / 2-micrometer circle DNA / Establishment of Sister Chromatid Cohesion / Resolution of Sister Chromatid Cohesion / tRNA gene clustering / meiotic cohesin complex / cohesin loader activity / establishment of meiotic sister chromatid cohesion / DNA secondary structure binding / mitotic cohesin complex / cohesin complex / rDNA chromatin condensation / establishment of protein localization to chromatin / transcription-dependent tethering of RNA polymerase II gene DNA at nuclear periphery / synaptonemal complex assembly / SUMOylation of DNA damage response and repair proteins / meiotic sister chromatid cohesion / replication-born double-strand break repair via sister chromatid exchange / establishment of mitotic sister chromatid cohesion / mitotic chromosome condensation / reciprocal meiotic recombination / sister chromatid cohesion / mitotic sister chromatid cohesion / protein acetylation / minor groove of adenine-thymine-rich DNA binding / mitotic sister chromatid segregation / chromosome, centromeric region / protein localization to chromatin / condensed nuclear chromosome / double-strand break repair / regulation of gene expression / double-stranded DNA binding / sequence-specific DNA binding / cell division / apoptotic process / DNA damage response / chromatin binding / chromatin / protein kinase binding / ATP hydrolysis activity / mitochondrion / DNA binding / ATP binding / identical protein binding / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Lee B-G / Gonzalez Llamazares A | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2020 ジャーナル: Elife / 年: 2020タイトル: Transport of DNA within cohesin involves clamping on top of engaged heads by Scc2 and entrapment within the ring by Scc3. 著者: James E Collier / Byung-Gil Lee / Maurici Brunet Roig / Stanislav Yatskevich / Naomi J Petela / Jean Metson / Menelaos Voulgaris / Andres Gonzalez Llamazares / Jan Löwe / Kim A Nasmyth /  要旨: In addition to extruding DNA loops, cohesin entraps within its SMC-kleisin ring (S-K) individual DNAs during G1 and sister DNAs during S-phase. All three activities require related hook-shaped ...In addition to extruding DNA loops, cohesin entraps within its SMC-kleisin ring (S-K) individual DNAs during G1 and sister DNAs during S-phase. All three activities require related hook-shaped proteins called Scc2 and Scc3. Using thiol-specific crosslinking we provide rigorous proof of entrapment activity in vitro. Scc2 alone promotes entrapment of DNAs in the E-S and E-K compartments, between ATP-bound engaged heads and the SMC hinge and associated kleisin, respectively. This does not require ATP hydrolysis nor is it accompanied by entrapment within S-K rings, which is a slower process requiring Scc3. Cryo-EM reveals that DNAs transported into E-S/E-K compartments are 'clamped' in a sub-compartment created by Scc2's association with engaged heads whose coiled coils are folded around their elbow. We suggest that clamping may be a recurrent feature of cohesin complexes active in loop extrusion and that this conformation precedes the S-K entrapment required for sister chromatid cohesion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11585.map.gz emd_11585.map.gz | 8.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11585-v30.xml emd-11585-v30.xml emd-11585.xml emd-11585.xml | 27 KB 27 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11585.png emd_11585.png | 147.5 KB | ||
| マスクデータ |  emd_11585_msk_1.map emd_11585_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-11585.cif.gz emd-11585.cif.gz | 7.7 KB | ||
| その他 |  emd_11585_half_map_1.map.gz emd_11585_half_map_1.map.gz emd_11585_half_map_2.map.gz emd_11585_half_map_2.map.gz | 98.5 MB 98.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11585 http://ftp.pdbj.org/pub/emdb/structures/EMD-11585 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11585 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11585 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11585_validation.pdf.gz emd_11585_validation.pdf.gz | 708.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11585_full_validation.pdf.gz emd_11585_full_validation.pdf.gz | 707.8 KB | 表示 | |
| XML形式データ |  emd_11585_validation.xml.gz emd_11585_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_11585_validation.cif.gz emd_11585_validation.cif.gz | 16.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11585 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11585 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11585 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11585 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11585.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11585.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11585_msk_1.map emd_11585_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11585_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11585_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Complex of cohesin, Scc2, ATP and DNA
+超分子 #1: Complex of cohesin, Scc2, ATP and DNA
+超分子 #2: Complex of cohesin and Scc2
+超分子 #3: double strand DNA
+分子 #1: Structural maintenance of chromosomes protein 1,Structural mainte...
+分子 #2: Structural maintenance of chromosomes protein 3,Structural mainte...
+分子 #3: Sister chromatid cohesion protein 1,Sister chromatid cohesion pro...
+分子 #4: Sister chromatid cohesion protein 2
+分子 #5: DNA (34-MER)
+分子 #6: DNA (34-MER)
+分子 #7: ADENOSINE-5'-TRIPHOSPHATE
+分子 #8: MAGNESIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 100 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 588164 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X