+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11008 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Icosahedral reconstruction of human adenovirus D26 capsid | |||||||||
 マップデータ マップデータ | Icosahedral map of human adenovirus D26 capsid | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
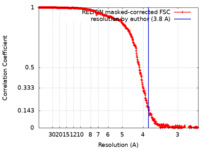
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Huiskonen JT / Abrishami V | |||||||||
| 資金援助 | European Union,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Prog Biophys Mol Biol / 年: 2021 ジャーナル: Prog Biophys Mol Biol / 年: 2021タイトル: Localized reconstruction in Scipion expedites the analysis of symmetry mismatches in cryo-EM data. 著者: Vahid Abrishami / Serban L Ilca / Josue Gomez-Blanco / Ilona Rissanen / José Miguel de la Rosa-Trevín / Vijay S Reddy / José-Maria Carazo / Juha T Huiskonen /       要旨: Technological advances in transmission electron microscopes and detectors have turned cryogenic electron microscopy (cryo-EM) into an essential tool for structural biology. A commonly used cryo-EM ...Technological advances in transmission electron microscopes and detectors have turned cryogenic electron microscopy (cryo-EM) into an essential tool for structural biology. A commonly used cryo-EM data analysis method, single particle analysis, averages hundreds of thousands of low-dose images of individual macromolecular complexes to determine a density map of the complex. The presence of symmetry in the complex is beneficial since each projection image can be assigned to multiple views of the complex. However, data processing that applies symmetry can average out asymmetric features and consequently data analysis methods are required to resolve asymmetric structural features. Scipion is a cryo-EM image processing framework that integrates functions from different image processing packages as plugins. To extend its functionality for handling symmetry mismatches, we present here a Scipion plugin termed LocalRec implementing the localized reconstruction method. When tested on an adenovirus data set, the plugin enables resolving the symmetry-mismatched trimeric fibre bound to the five-fold vertices of the capsid. Furthermore, it improves the structure determination of the icosahedral capsid by dealing with the defocus gradient across the particle. LocalRec is expected to be widely applicable in a range of cryo-EM investigations of flexible and symmetry mismatched complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11008.map.gz emd_11008.map.gz | 5.9 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11008-v30.xml emd-11008-v30.xml emd-11008.xml emd-11008.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11008_fsc.xml emd_11008_fsc.xml | 42.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11008.png emd_11008.png | 211.4 KB | ||
| マスクデータ |  emd_11008_msk_1.map emd_11008_msk_1.map | 6.4 GB |  マスクマップ マスクマップ | |
| その他 |  emd_11008_additional.map.gz emd_11008_additional.map.gz emd_11008_additional_1.map.gz emd_11008_additional_1.map.gz emd_11008_half_map_1.map.gz emd_11008_half_map_1.map.gz emd_11008_half_map_2.map.gz emd_11008_half_map_2.map.gz | 999.2 MB 999.2 MB 5.3 GB 5.3 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11008 http://ftp.pdbj.org/pub/emdb/structures/EMD-11008 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11008 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11008 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_11008_validation.pdf.gz emd_11008_validation.pdf.gz | 373.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_11008_full_validation.pdf.gz emd_11008_full_validation.pdf.gz | 372.7 KB | 表示 | |
| XML形式データ |  emd_11008_validation.xml.gz emd_11008_validation.xml.gz | 43 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11008 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11008 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11008 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11008 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 電子顕微鏡画像生データ |  EMPIAR-10455 (タイトル: Single particle cryo-EM dataset of human adenovirus HAdV-D26 EMPIAR-10455 (タイトル: Single particle cryo-EM dataset of human adenovirus HAdV-D26Data size: 46.5 Data #1: Particle stacks of human adenovirus HAdV-D26 [picked particles - single frame - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11008.map.gz / 形式: CCP4 / 大きさ: 6.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11008.map.gz / 形式: CCP4 / 大きさ: 6.4 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Icosahedral map of human adenovirus D26 capsid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
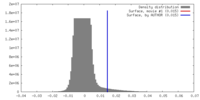
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_11008_msk_1.map emd_11008_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
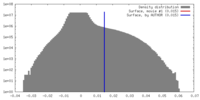
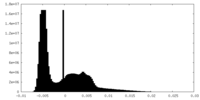
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_11008_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
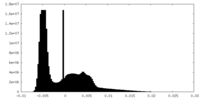
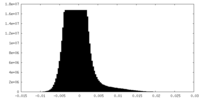
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_11008_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
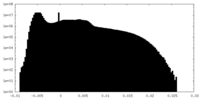
| 密度ヒストグラム |
-ハーフマップ: Half 1 of human adenovirus D26 capsid map
| ファイル | emd_11008_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half 1 of human adenovirus D26 capsid map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
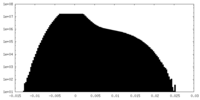
| 密度ヒストグラム |
-ハーフマップ: Half 2 of human adenovirus D26 capsid map
| ファイル | emd_11008_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half 2 of human adenovirus D26 capsid map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
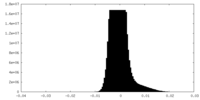
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Icosahedral reconstruction of human adenovirus D26
| 全体 | 名称: Icosahedral reconstruction of human adenovirus D26 |
|---|---|
| 要素 |
|
-超分子 #1: Icosahedral reconstruction of human adenovirus D26
| 超分子 | 名称: Icosahedral reconstruction of human adenovirus D26 / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-超分子 #2: A composite volume of human adenovirus D26 capsid created from lo...
| 超分子 | 名称: A composite volume of human adenovirus D26 capsid created from localized reconstructions of the four hexons タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293 Homo sapiens (ヒト) / 組換細胞: HEK293 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.1 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 7-38 / 実像数: 2000 / 平均露光時間: 7.6 sec. / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)