+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10109 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of beta-Galactosidase from Thermotoga maritima | |||||||||
 マップデータ マップデータ | Map from Relion3 Refine3D after postprocessing using the deposited mask and automatic filtering/sharpening, and sharpened with LocScale | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | BETA-GALACTOSIDASE / CARBOHYDRATE / GLUCOSYL HYDROLASE / THERMOTOGA MARITIMA / TRANSGLYCOSYLATION / IMMOBILIZATION / CRYOEM / GRAPHENE-OXIDE / GALACTOOLIGOSACCHARIDES / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lactose catabolic process / beta-galactosidase complex / beta-galactosidase / beta-galactosidase activity / carbohydrate binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Thermotoga maritima MSB8 (バクテリア) Thermotoga maritima MSB8 (バクテリア) | |||||||||
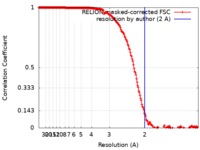
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.0 Å | |||||||||
 データ登録者 データ登録者 | Miguez-Amil S / Jimenez-Ortega E | |||||||||
 引用 引用 |  ジャーナル: ACS Chem Biol / 年: 2020 ジャーナル: ACS Chem Biol / 年: 2020タイトル: The cryo-EM Structure of β-Galactosidase: Quaternary Structure Guides Protein Engineering. 著者: Samuel Míguez Amil / Elena Jiménez-Ortega / Mercedes Ramírez-Escudero / David Talens-Perales / Julia Marín-Navarro / Julio Polaina / Julia Sanz-Aparicio / Rafael Fernandez-Leiro /  要旨: Lactose intolerance is a common digestive disorder that affects a large proportion of the adult human population. The severity of the symptoms is highly variable, depending on the susceptibility to ...Lactose intolerance is a common digestive disorder that affects a large proportion of the adult human population. The severity of the symptoms is highly variable, depending on the susceptibility to the sugar and the amount digested. For that reason, enzymes that can be used for the production of lactose-free milk and milk derivatives have acquired singular biotechnological importance. One such case is β-galactosidase (TmLac). Here, we report the cryo-EM structure of TmLac at 2.0 Å resolution. The protein features a newly solved domain at its C-terminus, characteristic of the genus , which promotes a peculiar octameric arrangement. We have assessed the constraints imposed by the quaternary protein structure on the construction of hybrid versions of this GH2 enzyme. Carbohydrate binding modules (CBM) from the CBM2 and CBM9 families have been added at either the amino or carboxy terminus, and the structural and functional effects of such modifications have been analyzed. The results provide a basis for the rational design of hybrid enzymes that can be efficiently attached to different solid supports. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10109.map.gz emd_10109.map.gz | 21.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10109-v30.xml emd-10109-v30.xml emd-10109.xml emd-10109.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10109_fsc.xml emd_10109_fsc.xml | 18.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10109.png emd_10109.png | 166 KB | ||
| マスクデータ |  emd_10109_msk_1.map emd_10109_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-10109.cif.gz emd-10109.cif.gz | 6.9 KB | ||
| その他 |  emd_10109_half_map_1.map.gz emd_10109_half_map_1.map.gz emd_10109_half_map_2.map.gz emd_10109_half_map_2.map.gz | 405.3 MB 405.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10109 http://ftp.pdbj.org/pub/emdb/structures/EMD-10109 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10109 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10109 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_10109_validation.pdf.gz emd_10109_validation.pdf.gz | 916 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_10109_full_validation.pdf.gz emd_10109_full_validation.pdf.gz | 915.6 KB | 表示 | |
| XML形式データ |  emd_10109_validation.xml.gz emd_10109_validation.xml.gz | 27 KB | 表示 | |
| CIF形式データ |  emd_10109_validation.cif.gz emd_10109_validation.cif.gz | 34.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10109 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10109 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10109 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-10109 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10109.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10109.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map from Relion3 Refine3D after postprocessing using the deposited mask and automatic filtering/sharpening, and sharpened with LocScale | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.67 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
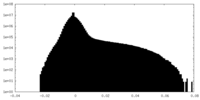
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10109_msk_1.map emd_10109_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
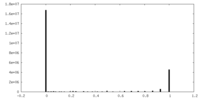
| 密度ヒストグラム |
-ハーフマップ: Half map 2 from Relion3
| ファイル | emd_10109_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 from Relion3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
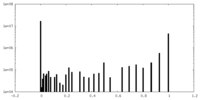
| 密度ヒストグラム |
-ハーフマップ: Half map 2 from Relion3
| ファイル | emd_10109_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 from Relion3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
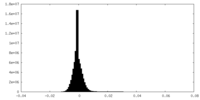
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Map of beta-Galactosidase from Thermotoga maritima
| 全体 | 名称: Map of beta-Galactosidase from Thermotoga maritima |
|---|---|
| 要素 |
|
-超分子 #1: Map of beta-Galactosidase from Thermotoga maritima
| 超分子 | 名称: Map of beta-Galactosidase from Thermotoga maritima / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Map from Relion3: Refined 3D after postprocessing using the deposited mask and automatic filtering/sharpening. Sharpened by LocScale |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima MSB8 (バクテリア) Thermotoga maritima MSB8 (バクテリア) |
| 分子量 | 理論値: 1.02 MDa |
-分子 #1: Beta-galactosidase
| 分子 | 名称: Beta-galactosidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: beta-galactosidase |
|---|---|
| 由来(天然) | 生物種:   Thermotoga maritima MSB8 (バクテリア) Thermotoga maritima MSB8 (バクテリア) |
| 分子量 | 理論値: 127.653539 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PYEWENPQLV SEGTEKSHAS FIPYLDPFSG EWEYPEEFIS LNGNWRFLFA KNPFEVPEDF FSEKFDDSNW DEIEVPSNWE MKGYGKPIY TNVVYPFEPN PPFVPKDDNP TGVYRRWIEI PEDWFKKEIF LHFEGVRSFF YLWVNGKKIG FSKDSCTPAE F RLTDVLRP ...文字列: PYEWENPQLV SEGTEKSHAS FIPYLDPFSG EWEYPEEFIS LNGNWRFLFA KNPFEVPEDF FSEKFDDSNW DEIEVPSNWE MKGYGKPIY TNVVYPFEPN PPFVPKDDNP TGVYRRWIEI PEDWFKKEIF LHFEGVRSFF YLWVNGKKIG FSKDSCTPAE F RLTDVLRP GKNLITVEVL KWSDGSYLED QDMWWFAGIY RDVYLYALPK FHIRDVFVRT DLDENYRNGK IFLDVEMRNL GE EEEKDLE VTLITPDGDE KTLVKETVKP EDRVLSFAFD VKDPKKWSAE TPHLYVLKLK LGEDEKKVNF GFRKIEIKDG TLL FNGKPL YIKGVNRHEF DPDRGHAVTV ERMIQDIKLM KQHNINTVRT SHYPNQTKWY DLCDYFGLYV IDEANIESHG IDWD PEVTL ANRWEWEKAH FDRIKRMVER DKNHPSIIFW SLGNEAGDGV NFEKAALWIK KRDNTRLIHY EGTTRRGESY YVDVF SLMY PKMDILLEYA SKKREKPFIM CEYAHAMGNS VGNLKDYWDV IEKYPYLHGG CIWDWVDQGI RKKDENGREF WAYGGD FGD TPNDGNFCIN GVVLPDRTPE PELYEVKKVY QNVKIRQVSK DTYEVENRYL FTNLEMFDGA WKIRKDGEVI EEKTFKI FA EPGEKRLLKI PLPEMDDSEY FLEISFSLSE DTPWAEKGHV VAWEQFLLKA PAFEKKSISD GVSLREDGKH LTVEAKDT V YVFSKLTGLL EQILHRRKKI LKSPVVPNFW RVPTDNDIGN RMPQRLAIWK RASKERKLFK MHWKKEENRV SVHSVFQLP GNSWVYTTYT VFGNGDVLVD LSLIPAEDVP EIPRIGFQFT VPEEFGTVEW YGRGPHETYW DRKESGLFAR YRKAVGEMMH RYVRPQETG NRSDVRWFAL SDGETKLFVS GMPQIDFSVW PFSMEDLERV QHISELPERD FVTVNVDFRQ MGLGGDDSWG A MPHLEYRL LPKPYRFSFR MRISEEIPSW RVLAAIPETL HVEMSSEDVI REGDTLRVKF SLLNDTPLSK EKQVVLFVDG NE YSVRRVV IPPFKKEELV FKVEGLKKGE HLIHTNLNTR KTIYVR UniProtKB: Beta-galactosidase |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 8 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 1384 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 30.0 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 3600 / 平均露光時間: 25.0 sec. / 平均電子線量: 35.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 2.6 µm / 最小 デフォーカス(補正後): 0.4 µm / 倍率(補正後): 20895 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 120000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-6s6z: |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)