| タイトル | Naturally Occurring Mutations of SARS-CoV-2 Main Protease Confer Drug Resistance to Nirmatrelvir. |
|---|
| ジャーナル・号・ページ | Acs Cent. Sci., Vol. 9, Page 1658-1669, Year 2023 |
|---|
| 掲載日 | 2022年6月17日 (構造データの登録日) |
|---|
 著者 著者 | Hu, Y. / Lewandowski, E.M. / Tan, H. / Zhang, X. / Morgan, R.T. / Jacobs, L.M.C. / Butler, S.G. / Gongora, M.V. / Choy, J. / Deng, X. ...Hu, Y. / Lewandowski, E.M. / Tan, H. / Zhang, X. / Morgan, R.T. / Jacobs, L.M.C. / Butler, S.G. / Gongora, M.V. / Choy, J. / Deng, X. / Chen, Y. / Wang, J. |
|---|
 リンク リンク |  Acs Cent. Sci. / Acs Cent. Sci. /  PubMed:37637734 PubMed:37637734 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.89 - 2.87 Å |
|---|
| 構造データ | PDB-8dcz:
Crystal Structure of SARS-CoV-2 Main Protease (Mpro) M165Y Mutant in Complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 2.38 Å PDB-8dd1:
SARS-CoV-2 Main Protease (Mpro) H164N Mutant in Complex with Inhibitor GC376
手法: X-RAY DIFFRACTION / 解像度: 2.03 Å PDB-8dd9:
Crystal Structure of SARS-CoV-2 Main Protease (Mpro) S144L Mutant in Complex with Inhibitor GC376
手法: X-RAY DIFFRACTION / 解像度: 2.04 Å PDB-8dfe:
Crystal Structure of SARS-CoV-2 Main Protease (Mpro) S144L Mutant
手法: X-RAY DIFFRACTION / 解像度: 1.89 Å PDB-8dfn:
Crystal Structure of SARS-CoV-2 Main Protease (Mpro) H164N Mutant
手法: X-RAY DIFFRACTION / 解像度: 2.04 Å PDB-8dgb:
Crystal Structure of SARS-CoV-2 Main Protease (Mpro) Q192T Mutant in Complex with Inhibitor GC376
手法: X-RAY DIFFRACTION / 解像度: 2.87 Å |
|---|
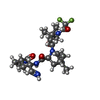
| 化合物 | ChemComp-4WI:
(1R,2S,5S)-N-{(1E,2S)-1-imino-3-[(3S)-2-oxopyrrolidin-3-yl]propan-2-yl}-6,6-dimethyl-3-[3-methyl-N-(trifluoroacetyl)-L-valyl]-3-azabicyclo[3.1.0]hexane-2-carboxamide / 薬剤, 抗ウイルス剤, プロテアーゼ阻害剤*YM /  ニルマトレルビル ニルマトレルビル
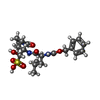
ChemComp-K36:
(1S,2S)-2-({N-[(benzyloxy)carbonyl]-L-leucyl}amino)-1-hydroxy-3-[(3S)-2-oxopyrrolidin-3-yl]propane-1-sulfonic acid / 薬剤, 抗ウイルス剤*YM /  GC376 GC376
ChemComp-B1S:
(1R,2S)-2-({N-[(benzyloxy)carbonyl]-L-leucyl}amino)-1-hydroxy-3-[(3S)-2-oxopyrrolidin-3-yl]propane-1-sulfonic acid
|
|---|
| 由来 |    severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2)
|
|---|
 キーワード キーワード |  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  Protease (プロテアーゼ) / Protease (プロテアーゼ) /  SARS-CoV-2 (SARSコロナウイルス2) / Mpro / SARS-CoV-2 (SARSコロナウイルス2) / Mpro /  Mutation (突然変異) Mutation (突然変異) |
|---|
 著者
著者 リンク
リンク Acs Cent. Sci. /
Acs Cent. Sci. /  PubMed:37637734
PubMed:37637734






 キーワード
キーワード VIRAL PROTEIN (ウイルスタンパク質) /
VIRAL PROTEIN (ウイルスタンパク質) /  Protease (プロテアーゼ) /
Protease (プロテアーゼ) /  SARS-CoV-2 (SARSコロナウイルス2) / Mpro /
SARS-CoV-2 (SARSコロナウイルス2) / Mpro /  Mutation (突然変異)
Mutation (突然変異) ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




