| タイトル | A computationally designed binding mode flip leads to a novel class of potent tri-vector cyclophilin inhibitors. |
|---|
| ジャーナル・号・ページ | Chem Sci, Vol. 10, Page 542-547, Year 2019 |
|---|
| 掲載日 | 2018年5月16日 (構造データの登録日) |
|---|
 著者 著者 | De Simone, A. / Georgiou, C. / Ioannidis, H. / Gupta, A.A. / Juarez-Jimenez, J. / Doughty-Shenton, D. / Blackburn, E.A. / Wear, M.A. / Richards, J.P. / Barlow, P.N. ...De Simone, A. / Georgiou, C. / Ioannidis, H. / Gupta, A.A. / Juarez-Jimenez, J. / Doughty-Shenton, D. / Blackburn, E.A. / Wear, M.A. / Richards, J.P. / Barlow, P.N. / Carragher, N. / Walkinshaw, M.D. / Hulme, A.N. / Michel, J. |
|---|
 リンク リンク |  Chem Sci / Chem Sci /  PubMed:30746096 PubMed:30746096 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.16 - 1.94 Å |
|---|
| 構造データ | PDB-6gji:
Cyclophilin A complexed with the tri-vector ligand 8.
手法: X-RAY DIFFRACTION / 解像度: 1.6 Å PDB-6gjj:
Cyclophilin A complexed with tri-vector ligand 2.
手法: X-RAY DIFFRACTION / 解像度: 1.38 Å PDB-6gjl:
Cyclophilin A complexed with tri-vector ligand 10.
手法: X-RAY DIFFRACTION / 解像度: 1.16 Å PDB-6gjm:
Cyclophilin A complexed with tri-vector ligand 4.
手法: X-RAY DIFFRACTION / 解像度: 1.354 Å PDB-6gjn:
Cyclophilin A complexed with tri-vector ligand 15.
手法: X-RAY DIFFRACTION / 解像度: 1.7 Å PDB-6gjp:
Cyclophilin A complexed with tri-vector ligand 7.
手法: X-RAY DIFFRACTION / 解像度: 1.94 Å PDB-6gjr:
Cyclophilin A complexed with tri-vector ligand 9.
手法: X-RAY DIFFRACTION / 解像度: 1.69 Å PDB-6gjy:
Cyclophilin A complexed with tri-vector ligand 5.
手法: X-RAY DIFFRACTION / 解像度: 1.29 Å |
|---|
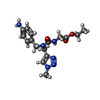
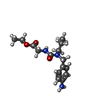
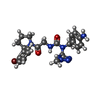
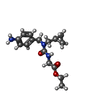
| 化合物 | ChemComp-F1E:
ethyl 2-[[(4-aminophenyl)methyl-[(1-methyl-1,2,3-triazol-4-yl)methyl]carbamoyl]amino]ethanoate
ChemComp-L36:
ethyl 2-[[(4-aminophenyl)methyl-propyl-carbamoyl]amino]ethanoate
ChemComp-F1Q:
ethyl 2-[[(4-aminophenyl)methyl-(1~{H}-1,2,3,4-tetrazol-5-ylmethyl)carbamoyl]amino]ethanoate
ChemComp-F0Q:
1-[(4-aminophenyl)methyl]-3-[2-[(2~{R})-2-(2-bromophenyl)pyrrolidin-1-yl]-2-oxidanylidene-ethyl]-1-[(2-methyl-1,2,3,4-tetrazol-5-yl)methyl]urea
ChemComp-F0T:
ethyl 2-[[(4-aminophenyl)methyl-(cyclopropylmethyl)carbamoyl]amino]ethanoate
ChemComp-F0W:
ethyl 2-[[(4-aminophenyl)methyl-[(2-methyl-1,2,3,4-tetrazol-5-yl)methyl]carbamoyl]amino]ethanoate
ChemComp-F1Z:
ethyl 2-[[(4-aminophenyl)methyl-prop-2-ynyl-carbamoyl]amino]ethanoate
|
|---|
| 由来 |  homo sapiens (ヒト) homo sapiens (ヒト)
|
|---|
 キーワード キーワード | ISOMERASE / Cyclophilins / CypA / inhibitor / PPIase / complex |
|---|
 著者
著者 リンク
リンク Chem Sci /
Chem Sci /  PubMed:30746096
PubMed:30746096












 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)