+Search query
-Structure paper
| Title | Discovery of a Potent Boronic Acid Derived Inhibitor of the HCV RNA-Dependent RNA Polymerase. |
|---|---|
| Journal, issue, pages | J. Med. Chem., Vol. 57, Page 1902-1913, Year 2014 |
| Publish date | Apr 22, 2013 (structure data deposition date) |
 Authors Authors | Maynard, A. / Crosby, R.M. / Ellis, B. / Hamatake, R. / Hong, Z. / Johns, B.A. / Kahler, K.M. / Koble, C. / Leivers, A. / Leivers, M.R. ...Maynard, A. / Crosby, R.M. / Ellis, B. / Hamatake, R. / Hong, Z. / Johns, B.A. / Kahler, K.M. / Koble, C. / Leivers, A. / Leivers, M.R. / Mathis, A. / Peat, A.J. / Pouliot, J.J. / Roberts, C.D. / Samano, V. / Schmidt, R.M. / Smith, G.K. / Spaltenstein, A. / Stewart, E.L. / Thommes, P. / Turner, E.M. / Voitenleitner, C. / Walker, J.T. / Waitt, G. / Weatherhead, J. / Weaver, K. / Williams, S. / Wright, L. / Xiong, Z.Z. / Haigh, D. / Shotwell, J.B. |
 External links External links |  J. Med. Chem. / J. Med. Chem. /  PubMed:23672667 PubMed:23672667 |
| Methods | X-ray diffraction |
| Resolution | 1.7 - 2.45 Å |
| Structure data |  PDB-4kai:  PDB-4kb7:  PDB-4kbi:  PDB-4ke5:  PDB-4khm:  PDB-4khr: |
| Chemicals | 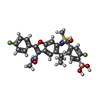 ChemComp-1PV:  ChemComp-HOH: 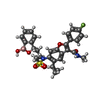 ChemComp-690: 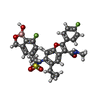 ChemComp-1C0:  ChemComp-SO4: |
| Source |
|
 Keywords Keywords | REPLICATION/REPLICATION INHIBITOR / HCV Polymerase / HCV NS5B / Site IV Inhibitor / boron / RNA Dependent RNA Polymerase / TRANSFERASE-TRANSFERASE INHIBITOR complex / REPLICATION-REPLICATION INHIBITOR complex / P66 / P70 / RNA directed RNA Polymerase / tar7360 / RNA-dependent RNA polymerase / Polymerase / RNA Replication / RNS Dependent RNA Polymerase |
 Movie
Movie Controller
Controller Structure viewers
Structure viewers About Yorodumi Papers
About Yorodumi Papers



 hepatitis c virus
hepatitis c virus