+検索条件
-Structure paper
| タイトル | The mechanism of STING autoinhibition and activation. |
|---|---|
| ジャーナル・号・ページ | Mol Cell, Vol. 83, Issue 9, Page 1502-1518.e10, Year 2023 |
| 掲載日 | 2023年5月4日 |
 著者 著者 | Sheng Liu / Bo Yang / Yingxiang Hou / Kaige Cui / Xiaozhu Yang / Xiaoxiong Li / Lianwan Chen / Shichao Liu / Zhichao Zhang / Yuanyuan Jia / Yufeng Xie / Ying Xue / Xiaomei Li / Bingxue Yan / Changxin Wu / Wen Deng / Jianxun Qi / Defen Lu / George F Gao / Peiyi Wang / Guijun Shang /  |
| PubMed 要旨 | 2',3'-cGAMP, produced by the DNA sensor cGAS, activates stimulator of interferon genes (STING) and triggers immune response during infection. Tremendous effort has been placed on unraveling the ...2',3'-cGAMP, produced by the DNA sensor cGAS, activates stimulator of interferon genes (STING) and triggers immune response during infection. Tremendous effort has been placed on unraveling the mechanism of STING activation. However, little is known about STING inhibition. Here, we found that apo-STING exhibits a bilayer with head-to-head as well as side-by-side packing, mediated by its ligand-binding domain (LBD). This type of assembly holds two endoplasmic reticulum (ER) membranes together not only to prevent STING ER exit but also to eliminate the recruitment of TBK1, representing the autoinhibited state of STING. Additionally, we obtained the filament structure of the STING/2',3'-cGAMP complex, which adopts a bent monolayer assembly mediated by LBD and transmembrane domain (TMD). The active, curved STING polymer could deform ER membrane to support its ER exit and anterograde transportation. Our data together provide a panoramic vision regarding STING autoinhibition and activation, which adds substantially to current understanding of the cGAS-STING pathway. |
 リンク リンク |  Mol Cell / Mol Cell /  PubMed:37086726 PubMed:37086726 |
| 手法 | EM (単粒子) |
| 解像度 | 3.3 Å |
| 構造データ | EMDB-35503, PDB-8ik0: EMDB-35504, PDB-8ik3: |
| 化合物 | 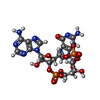 ChemComp-1SY: |
| 由来 |
|
 キーワード キーワード | IMMUNE SYSTEM / adaptor protein / complex |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について








 pseudomonas aeruginosa (strain atcc 15692 / dsm 22644 / cip 104116 / jcm 14847 / lmg 12228 / 1c / prs 101 / pao1) (緑膿菌)
pseudomonas aeruginosa (strain atcc 15692 / dsm 22644 / cip 104116 / jcm 14847 / lmg 12228 / 1c / prs 101 / pao1) (緑膿菌) homo sapiens (ヒト)
homo sapiens (ヒト)