| タイトル | Structural basis of nirmatrelvir and ensitrelvir activity against naturally occurring polymorphisms of the SARS-CoV-2 main protease. |
|---|
| ジャーナル・号・ページ | J. Biol. Chem., Vol. 299, Page 103004-103004, Year 2023 |
|---|
| 掲載日 | 2022年8月6日 (構造データの登録日) |
|---|
 著者 著者 | Noske, G.D. / de Souza Silva, E. / de Godoy, M.O. / Dolci, I. / Fernandes, R.S. / Guido, R.V.C. / Sjo, P. / Oliva, G. / Godoy, A.S. |
|---|
 リンク リンク |  J. Biol. Chem. / J. Biol. Chem. /  PubMed:36775130 PubMed:36775130 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.664 - 2.48 Å |
|---|
| 構造データ | PDB-8dz0:
Crystal Structure of SARS-CoV-2 Main protease in complex with Ensitrelvir
手法: X-RAY DIFFRACTION / 解像度: 2.29 Å PDB-8dz1:
Crystal Structure of SARS-CoV-2 Main protease mutant M49I in complex with Ensitrelvir
手法: X-RAY DIFFRACTION / 解像度: 2.08 Å PDB-8dz2:
Crystal Structure of SARS-CoV-2 Main protease in complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 2.129 Å PDB-8dz6:
Crystal Structure of SARS-CoV-2 Main protease mutant Q189K in complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 2.366 Å PDB-8dz9:
Crystal Structure of SARS-CoV-2 Main protease G143S mutant in complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 1.664 Å PDB-8dza:
Crystal Structure of SARS-CoV-2 Main protease A193T mutant in complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 1.961 Å PDB-8e1y:
Crystal Structure of SARS-CoV-2 Main protease A193S mutant in complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 2.48 Å PDB-8e25:
Crystal Structure of SARS-CoV-2 Main Protease M49I mutant in complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 1.868 Å PDB-8e26:
Crystal Structure of SARS-CoV-2 Main Protease N142S mutant in complex with Nirmatrelvir
手法: X-RAY DIFFRACTION / 解像度: 1.845 Å |
|---|
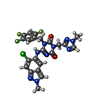
| 化合物 | ChemComp-7YY:
6-[(6-chloranyl-2-methyl-indazol-5-yl)amino]-3-[(1-methyl-1,2,4-triazol-3-yl)methyl]-1-[[2,4,5-tris(fluoranyl)phenyl]methyl]-1,3,5-triazine-2,4-dione / 薬剤, 抗ウイルス剤, プロテアーゼ阻害剤*YM
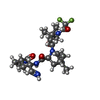
ChemComp-4WI:
(1R,2S,5S)-N-{(1E,2S)-1-imino-3-[(3S)-2-oxopyrrolidin-3-yl]propan-2-yl}-6,6-dimethyl-3-[3-methyl-N-(trifluoroacetyl)-L-valyl]-3-azabicyclo[3.1.0]hexane-2-carboxamide / 薬剤, 抗ウイルス剤, プロテアーゼ阻害剤*YM
|
|---|
| 由来 |   severe acute respiratory syndrome coronavirus 2 (ウイルス) severe acute respiratory syndrome coronavirus 2 (ウイルス)
|
|---|
 キーワード キーワード | VIRAL PROTEIN / HYDROLASE/INHIBITOR / Mpro / 3cl / covid / sars-cov-2 ensitrelvir / nirmatrelvir / HYDROLASE-INHIBITOR complex / Main Protease / SARS-CoV-2 |
|---|
 著者
著者 リンク
リンク J. Biol. Chem. /
J. Biol. Chem. /  PubMed:36775130
PubMed:36775130










 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




