+検索条件
-Structure paper
| タイトル | Structure and mechanism of a tripartite ATP-independent periplasmic TRAP transporter. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 14, Issue 1, Page 1120, Year 2023 |
| 掲載日 | 2023年2月27日 |
 著者 著者 | James S Davies / Michael J Currie / Rachel A North / Mariafrancesca Scalise / Joshua D Wright / Jack M Copping / Daniela M Remus / Ashutosh Gulati / Dustin R Morado / Sam A Jamieson / Michael C Newton-Vesty / Gayan S Abeysekera / Subramanian Ramaswamy / Rosmarie Friemann / Soichi Wakatsuki / Jane R Allison / Cesare Indiveri / David Drew / Peter D Mace / Renwick C J Dobson /      |
| PubMed 要旨 | In bacteria and archaea, tripartite ATP-independent periplasmic (TRAP) transporters uptake essential nutrients. TRAP transporters receive their substrates via a secreted soluble substrate-binding ...In bacteria and archaea, tripartite ATP-independent periplasmic (TRAP) transporters uptake essential nutrients. TRAP transporters receive their substrates via a secreted soluble substrate-binding protein. How a sodium ion-driven secondary active transporter is strictly coupled to a substrate-binding protein is poorly understood. Here we report the cryo-EM structure of the sialic acid TRAP transporter SiaQM from Photobacterium profundum at 2.97 Å resolution. SiaM comprises a "transport" domain and a "scaffold" domain, with the transport domain consisting of helical hairpins as seen in the sodium ion-coupled elevator transporter VcINDY. The SiaQ protein forms intimate contacts with SiaM to extend the size of the scaffold domain, suggesting that TRAP transporters may operate as monomers, rather than the typically observed oligomers for elevator-type transporters. We identify the Na and sialic acid binding sites in SiaM and demonstrate a strict dependence on the substrate-binding protein SiaP for uptake. We report the SiaP crystal structure that, together with docking studies, suggest the molecular basis for how sialic acid is delivered to the SiaQM transporter complex. We thus propose a model for substrate transport by TRAP proteins, which we describe herein as an 'elevator-with-an-operator' mechanism. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:36849793 / PubMed:36849793 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.04 - 3.03 Å |
| 構造データ | EMDB-13968, PDB-7qha: EMDB-15775, PDB-8b01:  PDB-7t3e: |
| 化合物 |  ChemComp-NA: 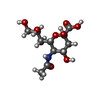 ChemComp-SLB:  ChemComp-SO4:  ChemComp-HOH: 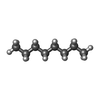 ChemComp-OCT:  ChemComp-PTY:  ChemComp-TWT:  ChemComp-D10:  ChemComp-TRD: 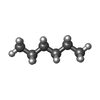 ChemComp-HEX: |
| 由来 |
|
 キーワード キーワード | TRANSPORT PROTEIN / TRAP / elevator / secondary transporter / sialic acid / Periplasmic SBP / TRAP transporter |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について







 photobacterium profundum (バクテリア)
photobacterium profundum (バクテリア)