+検索条件
-Structure paper
| タイトル | R-loop formation and conformational activation mechanisms of Cas9. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 609, Issue 7925, Page 191-196, Year 2022 |
| 掲載日 | 2022年8月24日 |
 著者 著者 | Martin Pacesa / Luuk Loeff / Irma Querques / Lena M Muckenfuss / Marta Sawicka / Martin Jinek /  |
| PubMed 要旨 | Cas9 is a CRISPR-associated endonuclease capable of RNA-guided, site-specific DNA cleavage. The programmable activity of Cas9 has been widely utilized for genome editing applications, yet its precise ...Cas9 is a CRISPR-associated endonuclease capable of RNA-guided, site-specific DNA cleavage. The programmable activity of Cas9 has been widely utilized for genome editing applications, yet its precise mechanisms of target DNA binding and off-target discrimination remain incompletely understood. Here we report a series of cryo-electron microscopy structures of Streptococcus pyogenes Cas9 capturing the directional process of target DNA hybridization. In the early phase of R-loop formation, the Cas9 REC2 and REC3 domains form a positively charged cleft that accommodates the distal end of the target DNA duplex. Guide-target hybridization past the seed region induces rearrangements of the REC2 and REC3 domains and relocation of the HNH nuclease domain to assume a catalytically incompetent checkpoint conformation. Completion of the guide-target heteroduplex triggers conformational activation of the HNH nuclease domain, enabled by distortion of the guide-target heteroduplex, and complementary REC2 and REC3 domain rearrangements. Together, these results establish a structural framework for target DNA-dependent activation of Cas9 that sheds light on its conformational checkpoint mechanism and may facilitate the development of novel Cas9 variants and guide RNA designs with enhanced specificity and activity. |
 リンク リンク |  Nature / Nature /  PubMed:36002571 / PubMed:36002571 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.54 - 4.14 Å |
| 構造データ | EMDB-14493, PDB-7z4c: EMDB-14494, PDB-7z4e: EMDB-14496, PDB-7z4g: EMDB-14497, PDB-7z4h: EMDB-14498, PDB-7z4i: EMDB-14499, PDB-7z4j: EMDB-14500, PDB-7z4k: EMDB-14501, PDB-7z4l:  PDB-7z4d: |
| 化合物 |  ChemComp-K:  ChemComp-HOH:  ChemComp-MG: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / CRISPR / Cas9 / R-loop / substrate binding / off-target |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について







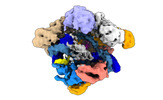

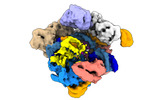



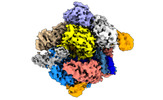

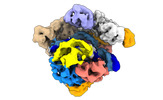



 streptococcus pyogenes (化膿レンサ球菌)
streptococcus pyogenes (化膿レンサ球菌)