+検索条件
-Structure paper
| タイトル | Structural basis for strychnine activation of human bitter taste receptor TAS2R46. |
|---|---|
| ジャーナル・号・ページ | Science, Vol. 377, Issue 6612, Page 1298-1304, Year 2022 |
| 掲載日 | 2022年9月16日 |
 著者 著者 | Weixiu Xu / Lijie Wu / Shenhui Liu / Xiao Liu / Xiaoling Cao / Cui Zhou / Jinyi Zhang / You Fu / Yu Guo / Yiran Wu / Qiwen Tan / Ling Wang / Junlin Liu / Longquan Jiang / Zhongbo Fan / Yuan Pei / Jingyi Yu / Jianjun Cheng / Suwen Zhao / Xiaojiang Hao / Zhi-Jie Liu / Tian Hua /  |
| PubMed 要旨 | Taste sensing is a sophisticated chemosensory process, and bitter taste perception is mediated by type 2 taste receptors (TAS2Rs), or class T G protein-coupled receptors. Understanding the detailed ...Taste sensing is a sophisticated chemosensory process, and bitter taste perception is mediated by type 2 taste receptors (TAS2Rs), or class T G protein-coupled receptors. Understanding the detailed molecular mechanisms behind taste sensation is hindered by a lack of experimental receptor structures. Here, we report the cryo-electron microscopy structures of human TAS2R46 complexed with chimeric mini-G protein gustducin, in both strychnine-bound and apo forms. Several features of TAS2R46 are disclosed, including distinct receptor structures that compare with known GPCRs, a new "toggle switch," activation-related motifs, and precoupling with mini-G protein gustducin. Furthermore, the dynamic extracellular and more-static intracellular parts of TAS2R46 suggest possible diverse ligand-recognition and activation processes. This study provides a basis for further exploration of other bitter taste receptors and their therapeutic applications. |
 リンク リンク |  Science / Science /  PubMed:36108005 PubMed:36108005 |
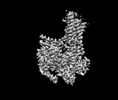
| 手法 | EM (単粒子) |
| 解像度 | 3.01 - 3.08 Å |
| 構造データ | EMDB-33364, PDB-7xp4: EMDB-33365, PDB-7xp5: EMDB-33366, PDB-7xp6: |
| 化合物 | 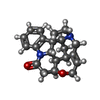 ChemComp-SY9: |
| 由来 |
|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  G protein-coupled receptor (Gタンパク質共役受容体) / taste type 2 receptors / cryo-electron microscopy structure G protein-coupled receptor (Gタンパク質共役受容体) / taste type 2 receptors / cryo-electron microscopy structure |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について