+検索条件
-Structure paper
| タイトル | Structural basis of the strict specificity of a bacterial GH31 α-1,3-glucosidase for nigerooligosaccharides. |
|---|---|
| ジャーナル・号・ページ | J Biol Chem, Vol. 298, Issue 5, Page 101827, Year 2022 |
| 掲載日 | 2022年3月12日 |
 著者 著者 | Marina Ikegaya / Toshio Moriya / Naruhiko Adachi / Masato Kawasaki / Enoch Y Park / Takatsugu Miyazaki /  |
| PubMed 要旨 | Carbohydrate-active enzymes are involved in the degradation, biosynthesis, and modification of carbohydrates and vary with the diversity of carbohydrates. The glycoside hydrolase (GH) family 31 is ...Carbohydrate-active enzymes are involved in the degradation, biosynthesis, and modification of carbohydrates and vary with the diversity of carbohydrates. The glycoside hydrolase (GH) family 31 is one of the most diverse families of carbohydrate-active enzymes, containing various enzymes that act on α-glycosides. However, the function of some GH31 groups remains unknown, as their enzymatic activity is difficult to estimate due to the low amino acid sequence similarity between characterized and uncharacterized members. Here, we performed a phylogenetic analysis and discovered a protein cluster (GH31_u1) sharing low sequence similarity with the reported GH31 enzymes. Within this cluster, we showed that a GH31_u1 protein from Lactococcus lactis (LlGH31_u1) and its fungal homolog demonstrated hydrolytic activities against nigerose [α-D-Glcp-(1→3)-D-Glc]. The k/K values of LlGH31_u1 against kojibiose and maltose were 13% and 2.1% of that against nigerose, indicating that LlGH31_u1 has a higher specificity to the α-1,3 linkage of nigerose than other characterized GH31 enzymes, including eukaryotic enzymes. Furthermore, the three-dimensional structures of LlGH31_u1 determined using X-ray crystallography and cryogenic electron microscopy revealed that LlGH31_u1 forms a hexamer and has a C-terminal domain comprising four α-helices, suggesting that it contributes to hexamerization. Finally, crystal structures in complex with nigerooligosaccharides and kojibiose along with mutational analysis revealed the active site residues involved in substrate recognition in this enzyme. This study reports the first structure of a bacterial GH31 α-1,3-glucosidase and provides new insight into the substrate specificity of GH31 enzymes and the physiological functions of bacterial and fungal GH31_u1 members. |
 リンク リンク |  J Biol Chem / J Biol Chem /  PubMed:35293315 / PubMed:35293315 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.75 - 2.73 Å |
| 構造データ | EMDB-32571, PDB-7wlg:  PDB-7wj9:  PDB-7wja:  PDB-7wjb:  PDB-7wjc:  PDB-7wjd:  PDB-7wje:  PDB-7wjf: |
| 化合物 |  ChemComp-XYL:  ChemComp-EDO:  ChemComp-HOH:  ChemComp-GLC:  ChemComp-BGC: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / nigerose / glucose / glycoside hydrolase / GH31 / carbohydrate / TIM-barrel / hexamer / kojibiose |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



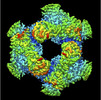

 lactococcus lactis subsp. cremoris mg1363 (乳酸菌)
lactococcus lactis subsp. cremoris mg1363 (乳酸菌)