+検索条件
-Structure paper
| タイトル | Structure of the NLRP3 decamer bound to the cytokine release inhibitor CRID3. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 604, Issue 7904, Page 184-189, Year 2022 |
| 掲載日 | 2022年2月3日 |
 著者 著者 | Inga V Hochheiser / Michael Pilsl / Gregor Hagelueken / Jonas Moecking / Michael Marleaux / Rebecca Brinkschulte / Eicke Latz / Christoph Engel / Matthias Geyer /  |
| PubMed 要旨 | NLRP3 is an intracellular sensor protein that when activated by a broad spectrum of exogenous and endogenous stimuli leads to inflammasome formation and pyroptosis. The conformational states of NLRP3 ...NLRP3 is an intracellular sensor protein that when activated by a broad spectrum of exogenous and endogenous stimuli leads to inflammasome formation and pyroptosis. The conformational states of NLRP3 and the way antagonistic small molecules act at the molecular level remain poorly understood. Here we report the cryo-electron microscopy structures of full-length human NLRP3 in its native form and complexed with the inhibitor CRID3 (also named MCC950). Inactive, ADP-bound NLRP3 is a decamer composed of homodimers of intertwined leucine-rich repeat (LRR) domains that assemble back-to-back as pentamers. The NACHT domain is located at the apical axis of this spherical structure. One pyrin domain dimer is in addition formed inside the LRR cage. Molecular contacts between the concave sites of two opposing LRR domains are mediated by an acidic loop that extends from an LRR transition segment. Binding of CRID3 considerably stabilizes the NACHT and LRR domains relative to each other. CRID3 binds into a cleft, connecting four subdomains of the NACHT with the transition LRR. Its central sulfonylurea group interacts with the Walker A motif of the NLRP3 nucleotide-binding domain and is sandwiched between two arginine residues, which explains the specificity of NLRP3 for this chemical entity. With the determination of the binding site of this key therapeutic agent, specific targeting of NLRP3 for the treatment of autoinflammatory and autoimmune diseases and rational drug optimization is within reach. |
 リンク リンク |  Nature / Nature /  PubMed:35114687 PubMed:35114687 |
| 手法 | EM (単粒子) |
| 解像度 | 3.84 - 9.6 Å |
| 構造データ |  EMDB-13684:  EMDB-13685: EMDB-13686: High resolution reconstruction of the human NLRP3 decamer  EMDB-13687:  EMDB-13692:  EMDB-13693:  EMDB-13699: |
| 化合物 |  ChemComp-ADP: 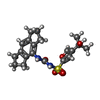 ChemComp-8GI: |
| 由来 |
|
 キーワード キーワード | IMMUNE SYSTEM / NLRP3 / CP-456.773 / CRID3 / MCC950 / AAA+ ATPase / NOD-like receptor / inflammasome |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





 homo sapiens (ヒト)
homo sapiens (ヒト)