| タイトル | STING cyclic dinucleotide sensing originated in bacteria. |
|---|
| ジャーナル・号・ページ | Nature, Vol. 586, Page 429-433, Year 2020 |
|---|
| 掲載日 | 2020年5月1日 (構造データの登録日) |
|---|
 著者 著者 | Morehouse, B.R. / Govande, A.A. / Millman, A. / Keszei, A.F.A. / Lowey, B. / Ofir, G. / Shao, S. / Sorek, R. / Kranzusch, P.J. |
|---|
 リンク リンク |  Nature / Nature /  PubMed:32877915 PubMed:32877915 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.52 - 2.9 Å |
|---|
| 構造データ | PDB-6wt4:
Structure of a bacterial STING receptor from Flavobacteriaceae sp. in complex with 3',3'-cGAMP
手法: X-RAY DIFFRACTION / 解像度: 1.78 Å PDB-6wt5:
Structure of a bacterial STING receptor from Capnocytophaga granulosa
手法: X-RAY DIFFRACTION / 解像度: 2.8 Å PDB-6wt6:
Structure of a metazoan TIR-STING receptor from C. gigas
手法: X-RAY DIFFRACTION / 解像度: 2.41 Å PDB-6wt7:
Structure of a metazoan TIR-STING receptor from C. gigas in complex with 2',3'-cGAMP
手法: X-RAY DIFFRACTION / 解像度: 2.9 Å PDB-6wt8:
Structure of a STING-associated CdnE c-di-GMP synthase from Flavobacteriaceae sp.
手法: X-RAY DIFFRACTION / 解像度: 1.52 Å PDB-6wt9:
Structure of STING-associated CdnE c-di-GMP synthase from Capnocytophaga granulosa
手法: X-RAY DIFFRACTION / 解像度: 2.3 Å |
|---|
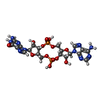
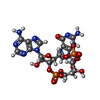
| 化合物 | ChemComp-4BW:
2-amino-9-[(2R,3R,3aS,5R,7aR,9R,10R,10aS,12R,14aR)-9-(6-amino-9H-purin-9-yl)-3,5,10,12-tetrahydroxy-5,12-dioxidooctahydro-2H,7H-difuro[3,2-d:3',2'-j][1,3,7,9,2,8]tetraoxadiphosphacyclododecin-2-yl]-1,9-dihydro-6H-purin-6-one / cGAMP
|
|---|
| 由来 |  flavobacteriaceae (バクテリア) flavobacteriaceae (バクテリア) capnocytophaga granulosa (バクテリア) capnocytophaga granulosa (バクテリア) crassostrea gigas (無脊椎動物) crassostrea gigas (無脊椎動物)
|
|---|
 キーワード キーワード | IMMUNE SYSTEM / CBASS / cyclic dinucleotide receptor / CD-NTase / immune effector / TIR domain / TRANSFERASE / c-di-GMP / STING |
|---|
 著者
著者 リンク
リンク Nature /
Nature /  PubMed:32877915
PubMed:32877915







 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 flavobacteriaceae (バクテリア)
flavobacteriaceae (バクテリア) crassostrea gigas (無脊椎動物)
crassostrea gigas (無脊椎動物)