+検索条件
-Structure paper
| タイトル | Structure of the neurotensin receptor 1 in complex with β-arrestin 1. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 579, Issue 7798, Page 303-308, Year 2020 |
| 掲載日 | 2020年1月16日 |
 著者 著者 | Weijiao Huang / Matthieu Masureel / Qianhui Qu / John Janetzko / Asuka Inoue / Hideaki E Kato / Michael J Robertson / Khanh C Nguyen / Jeffrey S Glenn / Georgios Skiniotis / Brian K Kobilka /   |
| PubMed 要旨 | Arrestin proteins bind to active, phosphorylated G-protein-coupled receptors (GPCRs), thereby preventing G-protein coupling, triggering receptor internalization and affecting various downstream ...Arrestin proteins bind to active, phosphorylated G-protein-coupled receptors (GPCRs), thereby preventing G-protein coupling, triggering receptor internalization and affecting various downstream signalling pathways. Although there is a wealth of structural information detailing the interactions between GPCRs and G proteins, less is known about how arrestins engage GPCRs. Here we report a cryo-electron microscopy structure of full-length human neurotensin receptor 1 (NTSR1) in complex with truncated human β-arrestin 1 (βarr1(ΔCT)). We find that phosphorylation of NTSR1 is critical for the formation of a stable complex with βarr1(ΔCT), and identify phosphorylated sites in both the third intracellular loop and the C terminus that may promote this interaction. In addition, we observe a phosphatidylinositol-4,5-bisphosphate molecule forming a bridge between the membrane side of NTSR1 transmembrane segments 1 and 4 and the C-lobe of arrestin. Compared with a structure of a rhodopsin-arrestin-1 complex, in our structure arrestin is rotated by approximately 85° relative to the receptor. These findings highlight both conserved aspects and plasticity among arrestin-receptor interactions. |
 リンク リンク |  Nature / Nature /  PubMed:31945771 / PubMed:31945771 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 4.2 Å |
| 構造データ | EMDB-20836, PDB-6up7: |
| 化合物 | 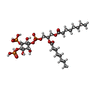 ChemComp-PIO: |
| 由来 |
|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / GPCR signaling MEMBRANE PROTEIN (膜タンパク質) / GPCR signaling |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について






