+検索条件
-Structure paper
| タイトル | Advances in long-wavelength native phasing at X-ray free-electron lasers. |
|---|---|
| ジャーナル・号・ページ | Iucrj, Vol. 7, Page 965-975, Year 2020 |
| 掲載日 | 2019年6月17日 (構造データの登録日) |
 著者 著者 | Nass, K. / Cheng, R. / Vera, L. / Mozzanica, A. / Redford, S. / Ozerov, D. / Basu, S. / James, D. / Knopp, G. / Cirelli, C. ...Nass, K. / Cheng, R. / Vera, L. / Mozzanica, A. / Redford, S. / Ozerov, D. / Basu, S. / James, D. / Knopp, G. / Cirelli, C. / Martiel, I. / Casadei, C. / Weinert, T. / Nogly, P. / Skopintsev, P. / Usov, I. / Leonarski, F. / Geng, T. / Rappas, M. / Dore, A.S. / Cooke, R. / Nasrollahi Shirazi, S. / Dworkowski, F. / Sharpe, M. / Olieric, N. / Bacellar, C. / Bohinc, R. / Steinmetz, M.O. / Schertler, G. / Abela, R. / Patthey, L. / Schmitt, B. / Hennig, M. / Standfuss, J. / Wang, M. / Milne, C.J. |
 リンク リンク |  Iucrj / Iucrj /  PubMed:33209311 PubMed:33209311 |
| 手法 | X線回折 |
| 解像度 | 1.95 - 2.65 Å |
| 構造データ |  PDB-6s0l:  PDB-6s0q:  PDB-6s19:  PDB-6s1d:  PDB-6s1e:  PDB-6s1g: |
| 化合物 | 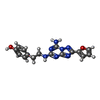 ChemComp-ZMA:  ChemComp-OLA: 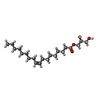 ChemComp-OLB:  ChemComp-OLC:  ChemComp-CLR:  ChemComp-TLA:  ChemComp-HOH: |
| 由来 |
|
 キーワード キーワード | SIGNALING PROTEIN / native-SAD / Serial femtosecond crystallography / SFX / SwissFEL / Jungfrau / MEMBRANE PROTEIN / PLANT PROTEIN |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)
 thaumatococcus daniellii (植物)
thaumatococcus daniellii (植物)