| 化合物 | ChemComp-RGY:
(4R,4aS,7aS,9S)-3,10-dimethyl-5,6,7,7a,8,9-hexahydro-4H-4a,9-epiminopyrrolo[3',4':5,6]cyclohepta[1,2-d][1,2]oxazol-4-ol
ChemComp-TQS:
(4R,4aR,7aS,9R)-3,10-dimethyl-5,6,7,7a,8,9-hexahydro-4H-4a,9-epiminopyrrolo[3',4':5,6]cyclohepta[1,2-d][1,2]oxazol-4-ol
ChemComp-RGV:
(3aS,8S,9aS)-10-methyl-4-oxo-1,4,6,8,9,9a-hexahydro-3a,8-epiminocyclohepta[1,2-c:4,5-c']dipyrrole-2(3H)-carbaldehyde
ChemComp-RGM:
(3aS,8S,9aS)-2-acetyl-10-methyl-2,3,6,8,9,9a-hexahydro-3a,8-epiminocyclohepta[1,2-c:4,5-c']dipyrrol-4(1H)-one
ChemComp-RGD:
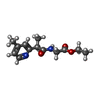
cyclobutyl[(2S,3aS,6aS)-6a-(2-hydroxyethyl)-2-(hydroxymethyl)hexahydro-5H-furo[2,3-c]pyrrol-5-yl]methanone
ChemComp-RHJ:
cyclobutyl[(3aS,4R,5R,7S,8aS)-4,5-dihydroxyhexahydro-1H-3a,7-epoxycyclohepta[c]pyrrol-2(3H)-yl]methanone
ChemComp-RHG:
(3aR,8S,9aS)-2-[(trifluoromethyl)sulfonyl]decahydro-3a,8-epoxypyrrolo[3,4-c]azocine
ChemComp-RH7:
methyl (5aS,8aS,10S)-8,8a,9,10-tetrahydro-5H-5a,10-epoxypyrrolo[3',4':4,5]cyclohepta[1,2-b]pyrazine-7(6H)-carboxylate
ChemComp-RH4:
methyl (2R)-3-(furan-2-yl)-2-(pyridin-4-yl)propanoate
ChemComp-RHV:
ethyl N-[(2R)-2-(4-methylpyridin-2-yl)propanoyl]glycinate
ChemComp-RHY:
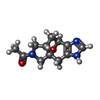
1-[(4aS,7aS,9S)-3,4,7,7a,8,9-hexahydro-4a,9-epoxypyrrolo[3',4':4,5]cyclohepta[1,2-d]imidazol-6(5H)-yl]ethan-1-one
ChemComp-RJ4:
(4aR,7aR,9R)-3,4,7,7a,8,9-hexahydro-4a,9-epoxypyrrolo[3',4':4,5]cyclohepta[1,2-d]imidazole-6(5H)-carbaldehyde
ChemComp-RJ7:
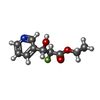
ethyl (2S,3S)-2-fluoro-3-hydroxy-3-(pyridin-3-yl)propanoate
ChemComp-RJD:
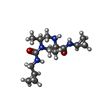
(3S,6S)-N~3~-cyclopropyl-N~1~-(cyclopropylmethyl)-3,6-dimethylpiperazine-1,3-dicarboxamide
ChemComp-RJG:
(3aR,10R,10aR,11aR)-1,2,3,5,10,10a,11,11a-octahydro-3a,10-ethanopyrrolo[3',4':4,5]pyrrolo[1,2-b]isoquinolin-13-one
ChemComp-RJJ:
(5R)-N-benzyl-5-ethyl-1-methyl-1,4-diazepane-5-carboxamide
ChemComp-RK1:
(7R)-N-benzyl-1,7-diethyl-2,3,6,7-tetrahydro-1H-1,4-diazepine-7-carboxamide
ChemComp-RK7:
ethyl (2R,3S)-3-(5-bromopyridin-2-yl)-2-fluoro-3-hydroxypropanoate
ChemComp-RKG:
methyl (4aS,7aS,9S)-3,4,7,7a,8,9-hexahydro-4a,9-epoxypyrrolo[3',4':4,5]cyclohepta[1,2-d]imidazole-6(5H)-carboxylate
ChemComp-RKJ:
(4R,4aS,7aS,9S)-6-ethyl-3,10-dimethyl-5,6,7,7a,8,9-hexahydro-4H-4a,9-epiminopyrrolo[3',4':5,6]cyclohepta[1,2-d][1,2]oxazol-4-ol
ChemComp-RQP:
(5R)-5-[(4-fluorophenyl)methyl]-5-(2-hydroxyethyl)-3-(2-methoxyethyl)imidazolidine-2,4-dione
|
|---|
 著者
著者 リンク
リンク

























 キーワード
キーワード ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 硫酸塩
硫酸塩
