+検索条件
-Structure paper
| タイトル | Small molecule AX-024 reduces T cell proliferation independently of CD3ε/Nck1 interaction, which is governed by a domain swap in the Nck1-SH3.1 domain. |
|---|---|
| ジャーナル・号・ページ | J. Biol. Chem., Vol. 295, Page 7849-7864, Year 2020 |
| 掲載日 | 2019年12月13日 (構造データの登録日) |
 著者 著者 | Richter, K. / Rufer, A.C. / Muller, M. / Burger, D. / Casagrande, F. / Grossenbacher, T. / Huber, S. / Hug, M.N. / Koldewey, P. / D'Osualdo, A. ...Richter, K. / Rufer, A.C. / Muller, M. / Burger, D. / Casagrande, F. / Grossenbacher, T. / Huber, S. / Hug, M.N. / Koldewey, P. / D'Osualdo, A. / Schlatter, D. / Stoll, T. / Rudolph, M.G. |
 リンク リンク |  J. Biol. Chem. / J. Biol. Chem. /  PubMed:32317279 PubMed:32317279 |
| 手法 | X線回折 |
| 解像度 | 0.93 - 1.816 Å |
| 構造データ | 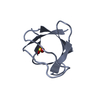 PDB-5qu1:  PDB-5qu2:  PDB-5qu3: 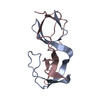 PDB-5qu4:  PDB-5qu5:  PDB-5qu6:  PDB-5qu7:  PDB-5qu8:  PDB-5qua: |
| 化合物 |  ChemComp-SO4:  ChemComp-HOH:  ChemComp-NA: |
| 由来 |
|
 キーワード キーワード | SIGNALING PROTEIN / SH3 DOMAIN / ADAPTOR / PEPTIDE BINDING / DOMAIN SWAP |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)