+検索条件
-Structure paper
| タイトル | GE23077 binds to the RNA polymerase 'i' and 'i+1' sites and prevents the binding of initiating nucleotides. |
|---|---|
| ジャーナル・号・ページ | Elife, Vol. 3, Page e02450-e02450, Year 2014 |
| 掲載日 | 2013年9月16日 (構造データの登録日) |
 著者 著者 | Zhang, Y. / Degen, D. / Ho, M.X. / Sineva, E. / Ebright, K.Y. / Ebright, Y.W. / Mekler, V. / Vahedian-Movahed, H. / Feng, Y. / Yin, R. ...Zhang, Y. / Degen, D. / Ho, M.X. / Sineva, E. / Ebright, K.Y. / Ebright, Y.W. / Mekler, V. / Vahedian-Movahed, H. / Feng, Y. / Yin, R. / Tuske, S. / Irschik, H. / Jansen, R. / Maffioli, S. / Donadio, S. / Arnold, E. / Ebright, R.H. |
 リンク リンク |  PubMed:24755292 PubMed:24755292 |
| 手法 | X線回折 |
| 解像度 | 2.8 - 3.624 Å |
| 構造データ |  PDB-4mq9:  PDB-4oin:  PDB-4oio:  PDB-4oip:  PDB-4oiq:  PDB-4oir: |
| 化合物 |  ChemComp-ZN:  ChemComp-MG:  ChemComp-MB8:  ChemComp-HOH:  ChemComp-ATP:  ChemComp-2TM: 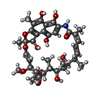 ChemComp-RFV: |
| 由来 |
|
 キーワード キーワード | TRANSCRIPTION / TRANSFERASE/ANTIBIOTIC / DNA-directed RNA polymerase / transferase-antibiotic complex / GE23077 / transcription inhibitor / transcription open complex / transcription initiation complex / i site / i+1 site / RNA polymerase / DNA/RNA/NTP binding / polymerization of ribonucleotides / nucleoid / TRANSFERASE / de novo transcription initiation / substrate complex / transcription initiation / CMPcPP / rifampicin / Rif / Rifampin / rifamycin SV |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 thermus thermophilus (バクテリア)
thermus thermophilus (バクテリア)