+検索条件
-Structure paper
| タイトル | Structures of human Golgi-resident glutaminyl cyclase and its complexes with inhibitors reveal a large loop movement upon inhibitor binding |
|---|---|
| ジャーナル・号・ページ | J. Biol. Chem., Vol. 286, Page 12439-12449, Year 2011 |
| 掲載日 | 2010年10月20日 (構造データの登録日) |
 著者 著者 | Huang, K.F. / Liaw, S.S. / Huang, W.L. / Chia, C.Y. / Lo, Y.C. / Chen, Y.L. / Wang, A.H.J. |
 リンク リンク |  J. Biol. Chem. / J. Biol. Chem. /  PubMed:21288892 PubMed:21288892 |
| 手法 | X線回折 |
| 解像度 | 1.05 - 1.95 Å |
| 構造データ |  PDB-3pb4:  PDB-3pb6:  PDB-3pb7:  PDB-3pb8:  PDB-3pb9:  PDB-3pbb:  PDB-3pbe: |
| 化合物 |  ChemComp-ZN:  ChemComp-HOH:  ChemComp-CAC: 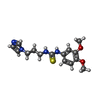 ChemComp-PBD: 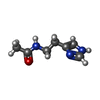 ChemComp-AHN: 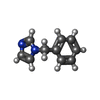 ChemComp-1BN:  ChemComp-SO4: |
| 由来 |
|
 キーワード キーワード | TRANSFERASE / alpha/beta protein / alpha/beta-mixed fold / glutaminyl cyclase / Golgi membrane / Transferase/Transferase inhibitor / Transferase-Transferase inhibitor complex / secretory pathway |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)