+検索条件
-Structure paper
| タイトル | Ligand-bound Structures Provide Atomic Snapshots for the Catalytic Mechanism of D-Amino Acid Deacylase |
|---|---|
| ジャーナル・号・ページ | J. Biol. Chem., Vol. 285, Page 5917-5930, Year 2010 |
| 掲載日 | 2009年11月12日 (構造データの登録日) |
 著者 著者 | Bhatt, T.K. / Yogavel, M. / Wydau, S. / Berwal, R. / Sharma, A. |
 リンク リンク |  J. Biol. Chem. / J. Biol. Chem. /  PubMed:20007323 PubMed:20007323 |
| 手法 | X線回折 |
| 解像度 | 2.09 - 3.3 Å |
| 構造データ |  PDB-3knf:  PDB-3knp:  PDB-3ko3:  PDB-3ko4:  PDB-3ko5:  PDB-3ko7:  PDB-3ko9:  PDB-3kob:  PDB-3koc:  PDB-3kod: |
| 化合物 |  ChemComp-ADP:  ChemComp-HOH: 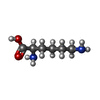 ChemComp-DLY:  ChemComp-DAR: 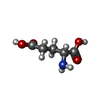 ChemComp-DGL:  ChemComp-DHI: 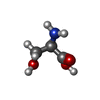 ChemComp-DSN: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / D-AMINO ACID / DEACYLASE / DTD / ADP / D-LYSINE / D-ARGININE / D-Glutamic acid / D-HISTIDINE / D-SERINE |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




