| タイトル | Docking for fragment inhibitors of AmpC beta-lactamase |
|---|
| ジャーナル・号・ページ | Proc. Natl. Acad. Sci. USA, Vol. 106, Page 7455-7460, Year 2009 |
|---|
| 掲載日 | 2009年3月24日 (構造データの登録日) |
|---|
 著者 著者 | Teotico, D.G. / Babaoglu, K. / Rocklin, G.J. / Ferreira, R.S. / Giannetti, A.M. / Shoichet, B.K. |
|---|
 リンク リンク |  Proc. Natl. Acad. Sci. USA / Proc. Natl. Acad. Sci. USA /  PubMed:19416920 PubMed:19416920 |
|---|
| 手法 | X線回折 |
|---|
| 解像度 | 1.8 - 2.49 Å |
|---|
| 構造データ | PDB-3gqz:
AmpC beta-lactamase in complex with Fragment-based Inhibitor
手法: X-RAY DIFFRACTION / 解像度: 1.8 Å PDB-3gr2:
AmpC beta-lactamase in complex with Fragment-based Inhibitor
手法: X-RAY DIFFRACTION / 解像度: 1.8 Å PDB-3grj:
AmpC beta-lactamase in complex with Fragment-based Inhibitor
手法: X-RAY DIFFRACTION / 解像度: 2.49 Å PDB-3gsg:
AmpC beta-lactamase in complex with Fragment-based Inhibitor
手法: X-RAY DIFFRACTION / 解像度: 2.1 Å PDB-3gtc:
AmpC beta-lactamase in complex with Fragment-based Inhibitor
手法: X-RAY DIFFRACTION / 解像度: 1.9 Å PDB-3gv9:
AmpC beta-lactamase in complex with Fragment-based Inhibitor
手法: X-RAY DIFFRACTION / 解像度: 1.8 Å PDB-3gvb:
AmpC beta-lactamase in complex with Fragment-based Inhibitor
手法: X-RAY DIFFRACTION / 解像度: 1.8 Å |
|---|
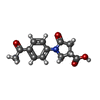
| 化合物 | ChemComp-GF7:
(3S)-1-(4-acetylphenyl)-5-oxopyrrolidine-3-carboxylic acid
ChemComp-GF4:
4-ethyl-5-methyl-2-(1H-tetrazol-5-yl)-1,2-dihydro-3H-pyrazol-3-one
ChemComp-GF1:
(2S)-2-[(3aR,4R,7S,7aS)-1,3-dioxooctahydro-2H-4,7-methanoisoindol-2-yl]propanoic acid
ChemComp-GTC:
(1R,2S)-2-(5-thioxo-4,5-dihydro-1H-1,2,4-triazol-3-yl)cyclohexanecarboxylic acid
ChemComp-GV9:
3-(acetylamino)thiophene-2-carboxylic acid
ChemComp-3GV:
(3S)-1-(2-hydroxyphenyl)-5-oxopyrrolidine-3-carboxylic acid
|
|---|
| 由来 | |
|---|
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR /  Hydrolase (加水分解酵素) / Hydrolase (加水分解酵素) /  Lactamase (Β-ラクタマーゼ) / Fragment / Lactamase (Β-ラクタマーゼ) / Fragment /  Antibiotic resistance (抗微生物薬耐性) / Antibiotic resistance (抗微生物薬耐性) /  Periplasm (ペリプラズム) / HYDROLASE-HYDROLASE INHIBITOR COMPLEX / AmpC Periplasm (ペリプラズム) / HYDROLASE-HYDROLASE INHIBITOR COMPLEX / AmpC |
|---|
 著者
著者 リンク
リンク Proc. Natl. Acad. Sci. USA /
Proc. Natl. Acad. Sci. USA /  PubMed:19416920
PubMed:19416920











 キーワード
キーワード Hydrolase (加水分解酵素) /
Hydrolase (加水分解酵素) /  Lactamase (Β-ラクタマーゼ) / Fragment /
Lactamase (Β-ラクタマーゼ) / Fragment /  Antibiotic resistance (抗微生物薬耐性) /
Antibiotic resistance (抗微生物薬耐性) /  Periplasm (ペリプラズム) / HYDROLASE-HYDROLASE INHIBITOR COMPLEX / AmpC
Periplasm (ペリプラズム) / HYDROLASE-HYDROLASE INHIBITOR COMPLEX / AmpC ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




