+検索条件
-Structure paper
| タイトル | Biochemical, Structural, and Biological Evaluation of Tranylcypromine Derivatives as Inhibitors of Histone Demethylases Lsd1 and Lsd2. |
|---|---|
| ジャーナル・号・ページ | J. Am. Chem. Soc., Vol. 132, Page 6827-, Year 2010 |
| 掲載日 | 2010年3月31日 (構造データの登録日) |
 著者 著者 | Binda, C. / Valente, S. / Romanenghi, M. / Pilotto, S. / Cirilli, R. / Karytinos, A. / Ciossani, G. / Botrugno, O.A. / Forneris, F. / Tardugno, M. ...Binda, C. / Valente, S. / Romanenghi, M. / Pilotto, S. / Cirilli, R. / Karytinos, A. / Ciossani, G. / Botrugno, O.A. / Forneris, F. / Tardugno, M. / Edmondson, D.E. / Minucci, S. / Mattevi, A. / Mai, A. |
 リンク リンク |  J. Am. Chem. Soc. / J. Am. Chem. Soc. /  PubMed:20415477 PubMed:20415477 |
| 手法 | X線回折 |
| 解像度 | 3.1 - 3.3 Å |
| 構造データ |  PDB-2xaf:  PDB-2xag:  PDB-2xah:  PDB-2xaj:  PDB-2xaq:  PDB-2xas: |
| 化合物 |  ChemComp-FAD: 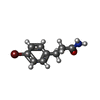 ChemComp-TCF: 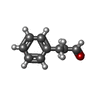 ChemComp-3PL: 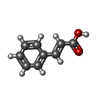 ChemComp-TCA:  ChemComp-M84:  ChemComp-M80: |
| 由来 |
|
 キーワード キーワード | TRANSCRIPTION / AMINE OXIDASE / CHROMATIN REGULATOR / HISTONE INHIBITOR BINDING / METHYLATION / NUCLEOSOME CORE / OXIDOREDUCTASE / OXIDOREDUCTASE-REPRESSOR COMPLEX / CHROMATIN REMODELLING |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 homo sapiens (ヒト)
homo sapiens (ヒト)