+検索条件
-Structure paper
| タイトル | Molecular structures of trimeric HIV-1 Env in complex with small antibody derivatives. |
|---|---|
| ジャーナル・号・ページ | Proc Natl Acad Sci U S A, Vol. 110, Issue 2, Page 513-518, Year 2013 |
| 掲載日 | 2013年1月8日 |
 著者 著者 | Joel R Meyerson / Erin E H Tran / Oleg Kuybeda / Weizao Chen / Dimiter S Dimitrov / Andrea Gorlani / Theo Verrips / Jeffrey D Lifson / Sriram Subramaniam /  |
| PubMed 要旨 | The extensive carbohydrate coat, the variability of protein structural features on HIV-1 envelope glycoproteins (Env), and the steric constraints of the virus-cell interface during infection, present ...The extensive carbohydrate coat, the variability of protein structural features on HIV-1 envelope glycoproteins (Env), and the steric constraints of the virus-cell interface during infection, present challenges to the elicitation of effective full-length (~150 kDa), neutralizing antibodies against HIV. These hurdles have motivated the engineering of smaller antibody derivatives that can bind Env and neutralize the virus. To further understand the mechanisms by which these proteins neutralize HIV-1, we carried out cryoelectron tomography of native HIV-1 BaL virions complexed separately to two small (~15 kDa) HIV-neutralizing proteins: A12, which binds the CD4-binding site on Env, and m36, whose binding to Env is enhanced by CD4 binding. We show that despite their small size, the presence of these proteins and their effects on the quaternary conformation of trimeric Env can be visualized in molecular structures derived by cryoelectron tomography combined with subvolume averaging. Binding of Env to A12 results in a conformational change that is comparable to changes observed upon its binding to the CD4-binding site antibody, b12. In contrast, binding of Env to m36 results in an "open" quaternary conformation similar to that seen with binding of soluble CD4 or the CD4i antibody, 17b. Because these small neutralizing proteins are less sterically hindered than full-length antibodies at zones of virus-cell contact, the finding that their binding has the same structural consequences as that of other broadly neutralizing antibodies highlights their potential for use in therapeutic applications. |
 リンク リンク |  Proc Natl Acad Sci U S A / Proc Natl Acad Sci U S A /  PubMed:23267106 / PubMed:23267106 /  PubMed Central PubMed Central |
| 手法 | EM (サブトモグラム平均) |
| 構造データ | 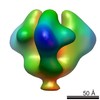 EMDB-5544:  EMDB-5551: 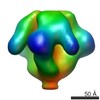 EMDB-5552:  EMDB-5553:  EMDB-5554:  EMDB-5555: |
| 由来 |
|
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)