+検索条件
-Structure paper
| タイトル | Tangled up in knots: structures of inactivated forms of E. coli class Ia ribonucleotide reductase. |
|---|---|
| ジャーナル・号・ページ | Structure, Vol. 20, Issue 8, Page 1374-1383, Year 2012 |
| 掲載日 | 2012年8月8日 |
 著者 著者 | Christina M Zimanyi / Nozomi Ando / Edward J Brignole / Francisco J Asturias / Joanne Stubbe / Catherine L Drennan /  |
| PubMed 要旨 | Ribonucleotide reductases (RNRs) provide the precursors for DNA biosynthesis and repair and are successful targets for anticancer drugs such as clofarabine and gemcitabine. Recently, we reported that ...Ribonucleotide reductases (RNRs) provide the precursors for DNA biosynthesis and repair and are successful targets for anticancer drugs such as clofarabine and gemcitabine. Recently, we reported that dATP inhibits E. coli class Ia RNR by driving formation of RNR subunits into α4β4 rings. Here, we present the first X-ray structure of a gemcitabine-inhibited E. coli RNR and show that the previously described α4β4 rings can interlock to form an unprecedented (α4β4)2 megacomplex. This complex is also seen in a higher-resolution dATP-inhibited RNR structure presented here, which employs a distinct crystal lattice from that observed in the gemcitabine-inhibited case. With few reported examples of protein catenanes, we use data from small-angle X-ray scattering and electron microscopy to both understand the solution conditions that contribute to concatenation in RNRs as well as present a mechanism for the formation of these unusual structures. |
 リンク リンク |  Structure / Structure /  PubMed:22727814 / PubMed:22727814 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 3.95 - 33.4 Å |
| 構造データ |  EMDB-5430: 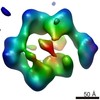 EMDB-5431:  EMDB-5432:  EMDB-5433: 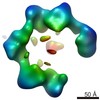 EMDB-5434:  EMDB-5435: 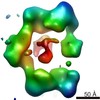 EMDB-5436:  EMDB-5437:  PDB-4erm:  PDB-4erp: |
| 化合物 |  ChemComp-DTP:  ChemComp-DAT:  ChemComp-MG:  ChemComp-FEO:  ChemComp-HOH:  ChemComp-ATP: |
| 由来 |
|
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / protein-protein complex / alpha/beta barrel / OXIDOREDUCTASE (酸化還元酵素) / protein-protein complex / alpha/beta barrel /  atp cone / di-iron center / RNR alpha / RNR beta / atp cone / di-iron center / RNR alpha / RNR beta /  thioredoxin (チオレドキシン) / Ribonucleotide reduction / thioredoxin (チオレドキシン) / Ribonucleotide reduction /  Cytosol (細胞質基質) / diiron center Cytosol (細胞質基質) / diiron center |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




