+検索条件
-Structure paper
| タイトル | nextPYP: a comprehensive and scalable platform for characterizing protein variability in situ using single-particle cryo-electron tomography. |
|---|---|
| ジャーナル・号・ページ | Nat Methods, Vol. 20, Issue 12, Page 1909-1919, Year 2023 |
| 掲載日 | 2023年10月26日 |
 著者 著者 | Hsuan-Fu Liu / Ye Zhou / Qinwen Huang / Jonathan Piland / Weisheng Jin / Justin Mandel / Xiaochen Du / Jeffrey Martin / Alberto Bartesaghi /  |
| PubMed 要旨 | Single-particle cryo-electron tomography is an emerging technique capable of determining the structure of proteins imaged within the native context of cells at molecular resolution. While high- ...Single-particle cryo-electron tomography is an emerging technique capable of determining the structure of proteins imaged within the native context of cells at molecular resolution. While high-throughput techniques for sample preparation and tilt-series acquisition are beginning to provide sufficient data to allow structural studies of proteins at physiological concentrations, the complex data analysis pipeline and the demanding storage and computational requirements pose major barriers for the development and broader adoption of this technology. Here, we present a scalable, end-to-end framework for single-particle cryo-electron tomography data analysis from on-the-fly pre-processing of tilt series to high-resolution refinement and classification, which allows efficient analysis and visualization of datasets with hundreds of tilt series and hundreds of thousands of particles. We validate our approach using in vitro and cellular datasets, demonstrating its effectiveness at achieving high-resolution and revealing conformational heterogeneity in situ. The framework is made available through an intuitive and easy-to-use computer application, nextPYP ( http://nextpyp.app ). |
 リンク リンク |  Nat Methods / Nat Methods /  PubMed:37884796 / PubMed:37884796 /  PubMed Central PubMed Central |
| 手法 | EM (サブトモグラム平均) |
| 解像度 | 1.8 - 12.4 Å |
| 構造データ |  EMDB-41196: A 3.2 Angstrom structure of HIV-1 CA-SP1 from 5 tomograms in EMPIAR-10164 dataset by single-particle cryo-electron tomography  EMDB-41197: A 3 Angstrom structure of HIV-1 CA-SP1 from EMPIAR-10164 dataset by single-particle cryo-electron tomography  EMDB-41199: 1.8 Angstrom mouse heavy chain apoferritin from EMPIAR-11273 dataset by single-particle cryo-electron tomography 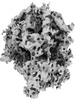 EMDB-41205: Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with P, A/T-site tRNA, and eEF1A cofactor from EMPIAR-10064 dataset by constrained classification  EMDB-41207: Cryo-electron tomography reconstruction of mammalian 80S ribosome from EMPIAR-10064 in the translocation intermediates (TI) state with eEF2 cofactor and closed L1 by constrained classification.  EMDB-41210: Cryo-electron tomography reconstruction of mammalian 80S ribosome in the translocation intermediates (TI) state with eEF2 cofactor and open L1 from EMPIAR-10064 dataset by constrained classification.  EMDB-41211: Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with P, A/T-site tRNA from EMPIAR-10064 dataset by constrained classification.  EMDB-41212: Cryo-electron tomography reconstruction of mammalian 80S ribosome in the non-rotated state with E, P, A/T-site tRNA and eEF1A cofactor from EMPIAR-10064 dataset by constrained classification.  EMDB-41220: Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in decoding state with EF-Tu-tRNA and P-site tRNA from EMPIAR-10499 dataset by constrained classification.  EMDB-41221: Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in classical non-rotated state with L1 open, obtained from EMPIAR-10499 dataset by constrained classification  EMDB-41222: Cryo-electron tomography reconstruction of M. pneumoniae 70S ribosome in the classical non-rotated state with half-closed L1 stalk, obtained from EMPIAR-10499 dataset by constrained classification  EMDB-41223: Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 1, obtained from the EMPIAR-10987 dataset by constrained 3D classification.  EMDB-41224: Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 2, obtained from the EMPIAR-10987 dataset by constrained 3D classification.  EMDB-41225: Cryo-electron tomography reconstruction of 80S ribosome in non-rotated state class 3, obtained from the EMPIAR-10987 dataset by constrained 3D classification. 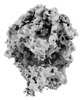 EMDB-41226: Cryo-electron tomography reconstruction of 80S ribosome in non-rotated decoding state class 4, obtained from the EMPIAR-10987 dataset by constrained 3D classification.  EMDB-41227: Cryo-electron tomography reconstruction of 80S ribosome in Translocation-intermediate-POST-3 state class 5, obtained from the EMPIAR-10987 dataset by constrained 3D classification. |
| 由来 |
|
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 Mycoplasmoides pneumoniae 19294 (バクテリア)
Mycoplasmoides pneumoniae 19294 (バクテリア)