+検索条件
-Structure paper
| タイトル | Structure of a functional obligate complex IIIIV respiratory supercomplex from Mycobacterium smegmatis. |
|---|---|
| ジャーナル・号・ページ | Nat Struct Mol Biol, Vol. 25, Issue 12, Page 1128-1136, Year 2018 |
| 掲載日 | 2018年12月5日 |
 著者 著者 | Benjamin Wiseman / Ram Gopal Nitharwal / Olga Fedotovskaya / Jacob Schäfer / Hui Guo / Qie Kuang / Samir Benlekbir / Dan Sjöstrand / Pia Ädelroth / John L Rubinstein / Peter Brzezinski / Martin Högbom /    |
| PubMed 要旨 | In the mycobacterial electron-transport chain, respiratory complex III passes electrons from menaquinol to complex IV, which in turn reduces oxygen, the terminal acceptor. Electron transfer is ...In the mycobacterial electron-transport chain, respiratory complex III passes electrons from menaquinol to complex IV, which in turn reduces oxygen, the terminal acceptor. Electron transfer is coupled to transmembrane proton translocation, thus establishing the electrochemical proton gradient that drives ATP synthesis. We isolated, biochemically characterized, and determined the structure of the obligate IIIIV supercomplex from Mycobacterium smegmatis, a model for Mycobacterium tuberculosis. The supercomplex has quinol:O oxidoreductase activity without exogenous cytochrome c and includes a superoxide dismutase subunit that may detoxify reactive oxygen species produced during respiration. We found menaquinone bound in both the Q and Q sites of complex III. The complex III-intrinsic diheme cytochrome cc subunit, which functionally replaces both cytochrome c and soluble cytochrome c in canonical electron-transport chains, displays two conformations: one in which it provides a direct electronic link to complex IV and another in which it serves as an electrical switch interrupting the connection. |
 リンク リンク |  Nat Struct Mol Biol / Nat Struct Mol Biol /  PubMed:30518849 PubMed:30518849 |
| 手法 | EM (単粒子) |
| 解像度 | 3.3 Å |
| 構造データ | |
| 化合物 |  ChemComp-FES:  ChemComp-CDL: 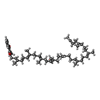 ChemComp-MQ9:  ChemComp-CU: 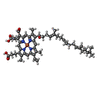 ChemComp-HAS:  ChemComp-HEC:  ChemComp-HEM: |
| 由来 |
|
 キーワード キーワード | ELECTRON TRANSPORT / Membrane Protein / Cryo-EM / Respiratory supercomplex / Mycobacterium |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について





 Mycobacterium smegmatis str. MC2 155 (バクテリア)
Mycobacterium smegmatis str. MC2 155 (バクテリア)