+Search query
-Structure paper
| Title | Structural Insights into Isovaleryl-Coenzyme A Dehydrogenase: Mechanisms of Substrate Specificity and Implications of Isovaleric Acidemia-Associated Mutations. |
|---|---|
| Journal, issue, pages | Research (Wash D C), Vol. 8, Page 0661, Year 2025 |
| Publish date | May 28, 2025 |
 Authors Authors | Kaide Ju / Fang Bai / Youwei Xu / Qingao Li / Gengchen Su / Ye Jin / Houzao Chen / Shuyang Zhang / Xiaodong Luan /  |
| PubMed Abstract | Isovaleryl-CoA (coenzyme A) dehydrogenase (IVD) plays a pivotal role in the catabolism of leucine, converting isovaleryl-CoA to 3-methylcrotonyl-CoA. Dysfunction of IVD is linked to isovaleric ...Isovaleryl-CoA (coenzyme A) dehydrogenase (IVD) plays a pivotal role in the catabolism of leucine, converting isovaleryl-CoA to 3-methylcrotonyl-CoA. Dysfunction of IVD is linked to isovaleric acidemia (IVA), a rare metabolic disorder characterized by the accumulation of toxic metabolites. In this study, we present the cryo-electron microscopy structures of human IVD, resolved both in its apo form and in complex with its substrates, isovaleryl-CoA and butyryl-CoA. Our findings reveal a tetrameric architecture with distinct substrate-binding pockets that facilitate the enzyme's preference for short branched-chain acyl-CoAs. Key residues involved in FAD binding and substrate interaction were identified, elucidating the catalytic mechanism of IVD. Additionally, we investigated the impact of various disease-associated hotspot mutations derived from different regions, demonstrating their effects on enzyme stability and activity. Notably, mutations such as A314V, S281G/F382V, and E411K resulted in substantial loss of function, while others exhibited milder effects, which is consistent with our structural analyses. These insights enhance our understanding of IVD's enzymatic properties and provide a foundation for developing targeted therapies for IVA. |
 External links External links |  Research (Wash D C) / Research (Wash D C) /  PubMed:40851894 / PubMed:40851894 /  PubMed Central PubMed Central |
| Methods | EM (single particle) |
| Resolution | 2.45 - 3.35 Å |
| Structure data | EMDB-61721, PDB-9jq3: EMDB-61722, PDB-9jq4: EMDB-61723, PDB-9jq5: |
| Chemicals |  ChemComp-FAD: 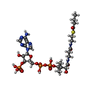 ChemComp-BCO: 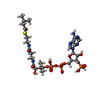 ChemComp-IVC: |
| Source |
|
 Keywords Keywords | HYDROLASE / IVD / FAD / butyryl-CoA / Isovaleryl-CoA |
 Movie
Movie Controller
Controller Structure viewers
Structure viewers About Yorodumi Papers
About Yorodumi Papers









 homo sapiens (human)
homo sapiens (human)