+検索条件
-Structure paper
| タイトル | A quinolin-8-ol sub-millimolar inhibitor of UGGT, the ER glycoprotein folding quality control checkpoint. |
|---|---|
| ジャーナル・号・ページ | Iscience, Vol. 26, Page 107919-107919, Year 2023 |
| 掲載日 | 2022年4月12日 (構造データの登録日) |
 著者 著者 | Guay, K.P. / Ibba, R. / Kiappes, J.L. / Vasiljevic, S. / Boni, F. / De Benedictis, M. / Zeni, I. / Le Cornu, J.D. / Hensen, M. / Chandran, A.V. ...Guay, K.P. / Ibba, R. / Kiappes, J.L. / Vasiljevic, S. / Boni, F. / De Benedictis, M. / Zeni, I. / Le Cornu, J.D. / Hensen, M. / Chandran, A.V. / Kantsadi, A.L. / Caputo, A.T. / Blanco Capurro, J.I. / Bayo, Y. / Hill, J.C. / Hudson, K. / Lia, A. / Brun, J. / Withers, S.G. / Marti, M. / Biasini, E. / Santino, A. / De Rosa, M. / Milani, M. / Modenutti, C.P. / Hebert, D.N. / Zitzmann, N. / Roversi, P. |
 リンク リンク |  Iscience / Iscience /  PubMed:37822503 PubMed:37822503 |
| 手法 | X線回折 |
| 解像度 | 1.649 - 2.049 Å |
| 構造データ |  PDB-7zkc:  PDB-7zll:  PDB-7zlu: |
| 化合物 |  ChemComp-CA:  ChemComp-HOH: 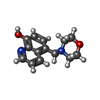 ChemComp-JM3:  ChemComp-NAG:  ChemComp-MAN: 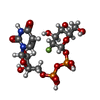 ChemComp-U2F: 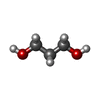 ChemComp-PDO: |
| 由来 |
|
 キーワード キーワード | TRANSFERASE / glycoprotein / misfolding / endoplasmic reticulum. UDP-glucose / GT24 / endoplasmic reticulum / 5-[(morpholin-4-yl)methyl]quinolin-8-ol / UDP-2-deoxy-2-fluoro-D-glucose |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



 chaetomium thermophilum (菌類)
chaetomium thermophilum (菌類)