+検索条件
-Structure paper
| タイトル | Structural insights into the mechanism of overcoming Erm-mediated resistance by macrolides acting together with hygromycin-A. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 14, Page 4196-4196, Year 2023 |
| 掲載日 | 2022年12月1日 (構造データの登録日) |
 著者 著者 | Chen, C.W. / Leimer, N. / Syroegin, E.A. / Dunand, C. / Bulman, Z.P. / Lewis, K. / Polikanov, Y.S. / Svetlov, M.S. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:37452045 PubMed:37452045 |
| 手法 | X線回折 |
| 解像度 | 2.35 - 2.65 Å |
| 構造データ |  PDB-8fc1:  PDB-8fc2:  PDB-8fc3:  PDB-8fc4:  PDB-8fc5:  PDB-8fc6: |
| 化合物 |  ChemComp-MG: 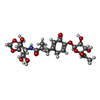 ChemComp-HGR: 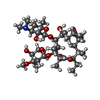 ChemComp-ERY:  ChemComp-MPD:  ChemComp-ARG:  ChemComp-ZN:  ChemComp-SF4:  ChemComp-HOH:  ChemComp-ZIT: 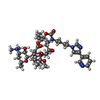 ChemComp-TEL: |
| 由来 |
|
 キーワード キーワード | RIBOSOME / Hygromycin A; macrolides; ketolides; erythromycin; azithromycin; telithromycin; antibiotic; synergy; Erm methyltransferase / X-ray structure; 70S ribosome; peptidyl transferase center; nascent peptide exit tunnel / Hygromycin A / macrolides / ketolides / erythromycin / azithromycin / telithromycin / antibiotic / synergy / Erm methyltransferase / 70S ribosome / peptidyl transferase center / nascent peptide exit tunnel |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




 thermus thermophilus hb8 (バクテリア)
thermus thermophilus hb8 (バクテリア)